Publicado:
2012-12-05Número:
Vol. 6 Núm. 2 (2012)Sección:
Visión InvestigadoraAnálisis computacional de proteínas de Helicobacter Pylori j99 y 26695
Palabras clave:
Helicobacter Pylori, bioinformática, proteína, método computacional. (es).Descargas
Referencias
R. Murillo, P. Pineros y G. Hernández Suarez, Atlas de mortalidad por cáncer en Colombia, Instituto Nacional de Cancerología e Instituto Geográfico Agustín Codazzi. Imprenta Nacional de Colombia, 2003.
R. Puffer y G. W. Griffith. “Cáncer,” en Características de la mortalidad urbana. Organización Panamericana de la Salud. Washington D. C.: Organización Mundial de la Salud, 1968.
A. Giraldo, “Tasa de mortalidad por cánceres del tubo digestivo según género y grupo de edad en Colombia entre 1980 y 1998,” Revista Colombiana de Gastroenterología,,vol. 23, no. 2: 124- 135, abril / junio 2008. [En línea]. Disponible en http://www.gastrocol.com/file/Revista/v23n2a05.pdf
IARC, Monographs on the evaluation of carcinogenic risks to humans. Schistosomes, liver flukes and Helicobacter pylori. Vol. 61. Lyon: International Agency for Research on Cancer, 1994.
M. Kidd y I. M. Modlin, A Century of Helicobacter Pylori: Paradigms Lost- Paradigms Regained. Digestion, vol. 59, no. 1, pp. 1-15. 1998.
F. Rivas, F. Hernández. Helicobacter pylori: Factores de virulencia, patología y diagnóstico. Rev Biomed; 11:187-205, 2000.
L. Bravo, “Helicobacter pylori: patología y prevalencia en biopsias gástricas en Colombia,” Colombia Médica, vol. 34, no. 3: 124-131, 2003. [En línea]. Disponible en http://redalyc.uaemex.mx/pdf/283/28334303.pdf
C. Pelayo, y B. Piazuelo, Cáncer gástrico: el enigma colombiano. Asociaciones Colombianas de Gastroenterología, Endoscopia digestiva, Coloproctología y Hepatología, Bogotá, 2010.
A. Alm, L Ling, D Moir, B King, et. al., “Genomic-sequence comparison of two unrelated isolates of the human gastric pathogen Helicobacter pylori,” Nature, vol. 367, no. 6715, pp. 176-180, 1999.
J. Tomb, O. White, A. Kerlavage, R. Clayton, vol. 338, no. 6642 et al., “The complete genome sequence of the gastric pathogen Helicobacter pylori,”. Nature, vol. 6642, no. 388, pp. 539-547, 1997.
W. R. Pearson, “Rapid and sensitive sequence comparison with FASTP and FASTA,” Methods Enzymol, vol. 183, pp. 63-98, 1990.
J. Kyte y R. F. Doolittle, “A simple method for displaying the hydropathic character of a protien,,” Journal of Molecular Biology, vol. 157, no. 1, pp. 105-132, mayo 1982.
T. P. Hoop y K. R. Woods, “Prediction of protein antigenic determinants from amino acid sequences,” Proc Natl Acad Sci, Vol. 78, no. 16, p. 3824, junio 1981.
D. Eisenberg, E. Schwarz, M. Komarony, Wall R Amino acid scale: Normalized consensus hydrophobicity scale. J. Mol. Biol.179:125-142. 1984
N. Blom, S. Gammeltoft y S. Brunak, “Sequence and structure-based prediction of eukaryotic protein phosphorylation sites,” Journal of Molecular Biology, vol. 294, no. 5, pp. 1351-1362, diciembre 1999.
K. Julenius, G. Molgaard, R. Gupta y S. Brunak, “Prediction, conservation analysis and structural characterización of mammalian mucin-type O-glycosylation sites,” Glycobiology, vol. 15, pp. 153-164. 2005.
N. Blon, T. Ponten, R. Gupta y S. Brunak S., “Prediction of post-translational glycosilation and phosphorylation of protein from aminoacid sequence,” Proteomics, vol. 4, pp. 1633-1649, 2004
J. Piotrowski, E. Piotrowski, D. Skrodzka, et. al., “Induction of acute gastritis an epithelial apoptosis by Helicobacter Pylori lipopolysaccharide,” Scand Gastroenterol. Vol. 32, no. 3, pp. 203- 211, 1997.
N. Rojas, El lipopolisacárido bacteriano: Una potente endotoxina con múltiples actividades biológicas. Costa Rica: Universidad de Costa Rica, 1995.
A. Lichtman, et. al., Inmunología celular y molecular. Madrid: Interamericana McGraw-Hill, 2000.
M. Nuñez, V. Scialom, D. Silva, et al., “Modelo teórico para una vacuna efectiva contra la infección por Helicobacter pylori,” Vitae, no. 28, 2006.
A. Marais, GL. Mendz, S. Hazell, F. Mégraud. Metabolism and Genetics of Helicobacter pylori: the Genome Era. Microbiology and Molecular Biology Reviews, vol. 63, no. 3, pp. 642-674, 1999.
R. A. Alm y B. Noonam, “The Genome,” en Helicobacter pylori: Physiology and Genetics. Washington, ASM Press, 2001.
H. Schlegel, Microbiología general. Barcelona, Editorial Omega. 1997.
R. K. Murray, P. A. Mayes, D. K. Granner y V. W. Rodwell, Bioquímica de Harper. México: Editorial Manual Moderno, 2001.
A. Lehninger, Bioquímica, Barcelona, Editorial Omega, 1995.
H. Kleanthous, G. A Myers y J. W. Ingrassia Rectal and intranasal immunizations with recombinant urease induce distinct local and serum immune responses in mice and protect against Helicobacter Pylori infection, Infection and Immunity, vol. 66, no. 6: 2879- 2886, junio 1998.
O. G Gómez, B. Lucas, Z. X. Yan, K. Panthel, T. F.Haas R., “Meyer. Protection of mice against gastrict colonization by Helicobacter Pylori by single oral dose immunization with attenuated Salmonella typhimurium producing urease subunits A and B,” Vaccine vol. 16, no.5: 460-471, marzo 1998.
M. Kawakubo, Y. Ito, M. Fukuda, et al., “Natural Antibiotic Function of a Human Gastric Mucin Against Helicobacter Pylori Infection,” Science, vol 305, no. 5686, pp. 1003-1006, agosto 2004.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas
Descargas
VISIÓN INVESTIGADORA
Visión Electrónica, 2012-12-01 Volumen:6, Año:2 pág: 58–81
ANÁLISIS COMPUTACIONAL DE PROTEÍNAS DE HELICOBACTER PYLORI J99 Y 26695
COMPUTATIONAL ANALYSIS OF PROTEINS (HELICOBACTER PYLORI J99 AND 26695)
Edilma E.Becerra
Ingeniera Electrónica de la Universidad Distrital Francisco José de Caldas, Facultad de Ingeniería. Correo electrónico: edilma.becerra@ gmail.com
Luz H. Camargo
Licenciada en Biología y Especialista en Bioingeniería. Candidata MSc. en Ingeniería Biomédica. Docente de la Facultad de Ingeniería de la Universidad Distrital Francisco José de Caldas y Directora del grupo de investigación INVID. Correo electrónico: lhcamargoc@udistrital. edu.co
RESUMEN
La colonización del Helicobacter Pylori en la mucosa gástrica es considerada como un adenocarcinoma debido a que su colonización e infección dentro de la mucosa gástrica influye de manera notable en el desarrollo del cáncer gástrico. Con el análisis del comportamiento eléctrico de las proteínas se pueden predecir interacciones electrostáticas que produzcan el posible encapsulamiento de la bacteria. En este trabajo se realizó un análisis computacional de cuatro proteínas de cada una de las cepas J99 y 26695, que inciden en la adhesión de la bacteria al epitelio gástrico y en el metabolismo, con ayuda de programas de libre acceso en Internet como Compute pI / Mw, Netphos 2.0, Net-Oglyc 3.1 y Net-Nglyc 1.0; para analizar los perfiles de hidrofobicidad se utilizó Protein Hidrophobicity Plot, como herramientas de comparación se utilizó Dot Plot y Dolet. El estudio revela entre 1 y 6 sitios potenciales de N-glicosilación en las proteínas estudiadas, en contraste con la ausencia de sitios potenciales de O-glicosilación e. Las características que debe cumplir la proteína de la familia escogida que encapsule a la bacteria son: punto isoeléctrico ser mayor a 7, con comportamiento eléctricamente positivo dentro del medio básico e hidrofílica. Se propone a las glucoproteínas N-glicosilisadas con radicales de N-acetilglucosamina como posible familia de proteínas encapsuladoras del Helicobacter Pylori.
Palabras claveHelicobacter Pylori, bioinformática, proteína, método computacional.
Abstract
The colonization of Helicobacter Pylori in gastric mucosa is considered an adenocarcinoma due to colonization and infection in the gastric mucosa significantly influence the development of gastric cancer. By analyzing the electrical behavior of proteins can be predicted electrostatic interactions that result from the possible encapsulation of the bacteria. In this work, a computational analysis of four proteins in each of the strains J99 and 26695, affecting the adhesion of bacteria to the gastric epithelium and metabolism, using freely available programs on the Internet and Compute pI / Mw, Netphen 2.0, 3.1 and Net-Net-Oglyc Nglyc 1.0, to analyze the hydrophobicity profiles Protein Hidrophobicity Plot was used as comparison tools used Dot Plot and Dolet. The study found between 1 and 6 potential sites for N-glycosylation in the proteins studied, in contrast to the lack of potential sites of O-glycosylation e. The features that should meet the chosen family protein that encapsulates the bacteria are isoelectric point be greater than 7, electrically positive behavior in the basic medium and hydrophilic. It is proposed that N-glycoproteins with radical glicosilisadas N-acetylglucosamine as a possible family of capping proteins of Helicobacter pylori.
Key WordsHelicobacter Pylori, bioinformatics, protein, computational method.
1. Introducción
El cáncer gástrico es la primera causa de muerte por cáncer en Colombia [1], [2], especialmente en los departamentos de Boyacá, Cundinamarca, Tolima y Santander [3]. En 1994 la Agencia Internacional para Investigación en Cáncer determino que la infección por Helicobacter Pylori (H. Pylori) era carcinogénica [4]. Se conoce que la infección H. Pylori induce una inflamación crónica en la mucosa del estómago, por lo cual ha sido identificada como la causa de gastritis aguda y crónica, ulcera péptica y gastritis atrófica que se intensifica por la respuesta del sistema inmunitario del huésped al liberar niveles elevados de citoquinas, lo que lleva a producir en su etapa final el cáncer gástrico [5].
Entre los factores de riesgo de infección por H. Pylori se encuentran la edad avanzada (mas de 60 años), el país de origen (los países tercermundistas tienen más probabilidades de tener un alto número de infectados), la posición socioeconómica (quienes viven en situación de hacinamiento, de pobreza, con familias numerosas y/o con menor higiene) e inadecuada alimentación, y existe prevalencia en las poblaciones de montaña [6], [7], [8].
Para combatir la infección de H. Pylori es necesario encontrar cadenas proteínicas capaces de encapsularla. Con ayuda de la bioinformática se puede acelerar este proceso de descubrimiento, lo que permite la rápida generación de una colección de candidatos potenciales para posteriores análisis en el laboratorio. El objetivo de este trabajo es realizar el análisis computacional de cuatro proteínas Helicobacter Pylori J99 Y 26695 y sugerir una familia de cadenas proteínicas biocompatibles con la bacteria, con base en su secuencia genómica para lograr su encapsulamiento utilizando las bases de datos biológicas existentes en Internet y las diferentes herramientas bioinformáticas que se encuentran actualmente.
2. Metodología
Análisis computacional de los genomas del H. Pylori J99 y 26695
El número de acceso para el genoma del H. Pylori J99 en NCBI es NC-000921, en Gen- Bank es AE001439 y en PubMed se ingresa con 9923682. El genoma completo de la bacteria fue reportado por Alm R y colaboradores [9], y el genoma del H. Pylori 26695, cuyo número de acceso en NCBI es NC-000915, en GenBank es AE000511 y en PubMed 9252185 fue reportado por Tomb y colaboradores [10]. Para realizar los análisis bioinformáticos la secuencia aminoacídica se descarga en formato FASTA [11].
El análisis computacional del punto isoeléctrico y el peso molecular se calcularon con Compute Pi / Mw. Los análisis de los perfiles de hidrofobicidad se realizaron con Protein Hidrophobicity Plot y las escalas de hidrofobicidad de Kyte-Doolittle [12] Hoop-Woods [13] y Eisenberg [14]. Los sitios potenciales de fosforilación se determinaron con Netphos 2.0 [15]; los de O-glicosilaciones, con el programa Net-Oglyc 3.1 [15], y los de N-glicosilaciones, con el programa Net-Nglyc 1.0 [16].
Como herramientas de comparación se utilizaron Dot Plot [17] y Dolet, de ExPasy y del Centro de Bioinformática del Instituto de Biotecnología de la Universidad Nacional de Colombia, respectivamente. Para determinar interacciones proteína-proteína de H. Pylori se utilizó la herramienta STRING 8.2 (Search Tool for the Retrieval of Interacting Genes/ Proteins).
3. Resultados y discusión
3.1. Análisis computacional de la estructura primaria, cuatro proteínas de H. Pylori J99
De 1408 proteínas codificadas para H. Pylori J99 se seleccionaron cuatro para el análisis computacional: el lipopolisacárido (LPS), la aminotransferasa fructosa-6-fosfato Glucosalina, la UDP-N-acetilglucosamina1-carboxivinil- transferasa y la Adhesina, considerando que estas proteínas están implicadas en la adhesión de la bacteria al epitelio gástrico y en su metabolismo.
3.1.1. Lipopolisacarido de H. Pylori J99
EL LPS de H. Pylori J99 es una contenida en la pared celular de las bacterias Gram negativas. La secuencia de aminoácidos puede ser consultada en GenBank con la referencia AAD06864 y en NCBI, con la referencia NP_224029. Esta proteína permite el reconocimiento taxonómico del organismo que lo posea y es importante en la especificación de antígenos en la bacteria; por tanto, es el responsable de la resistencia de la bacteria ante antimicrobianos [18]. Por otra parte, ha sido ampliamente estudiada en otros organismos bacterianos como Salmonella typhimurium y Escherichia coli [19], lo que ha permitido el desarrollo de medicamentos y vacunas [20]. La figura 1 muestra la secuencia de distribución de aminoácidos.
Figura 1. Lipopolisacarido de H. Pylori J99.
- Secuencia aminoacídica
- Histograma
de la distribución de aminoácidos
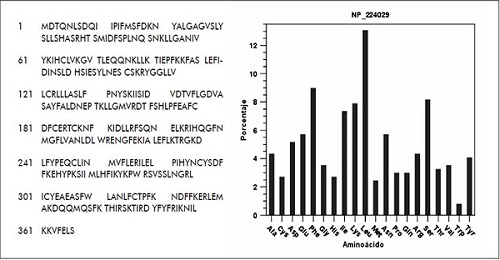
Fuente: elaboración propia.
Los valores de predicción computacional del punto isoeléctrico y peso molecular de la secuencia son 8,79 y 42,93 kDa, respectivamente, contienen 173 residuos hidrofóbicos y 99 residuos hidrofílicos. Se determinaron diez sitios potenciales de fosforilación, cuatro sitios potenciales de N-glicosilación, y ausencia de sitios potenciales de O-glicosilación (figura 2).
Figura 2. Lipopolisacárido de H. Pylori J99.
- Perfiles de hidrofobicidad.
- Sitios potenciales de fosforilación.
- Sitios potenciales de N-glicosilación
- Sitios potenciales de O-glicosilación
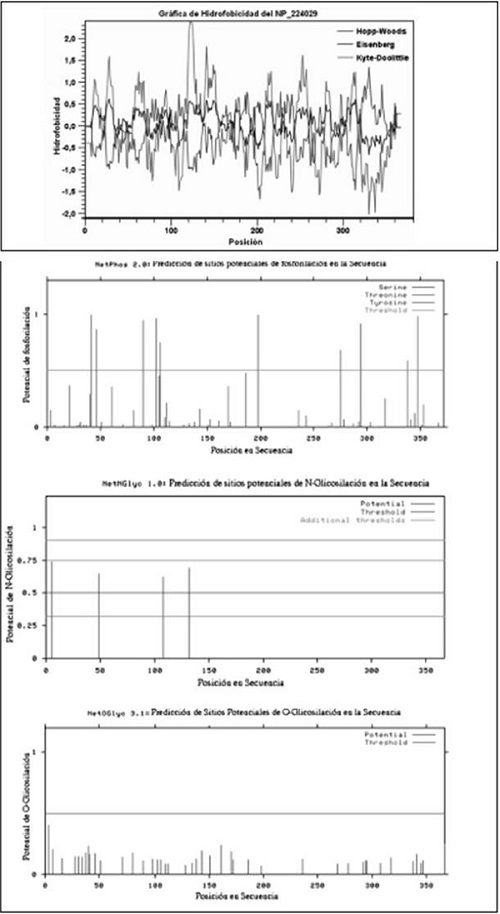
Fuente: elaboración propia.
3.1.2. Aminotransferasa fructosa-6-fosfato Glucosamina (glmS) H. Pylori J99
Esta proteína hace parte de una de las rutas metabólicas más importantes del H. Pylori J99, y permite así la transferencia de grupos amino, de un metabolito a otro. La secuencia de aminoácidos para glmS de H. Pylori J99 puede ser consultada en GenBank con la referencia AAD06999 y en NCBI, con la referencia NP_224138. La secuencia y distribución de aminoácidos se aprecia en la figura 3.
Figura 3. Aminotrasferasa fructosa-6-fosfato glucosamina de H. Pylori J99.
- Secuencia aminoacídica.
- Histograma de la distribución de aminoácidos
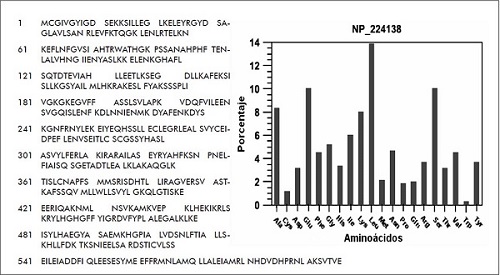
Fuente: elaboración propia.
Los valores de predicción computacional del punto isoeléctrico y el peso molecular de la secuencia son 6,39 y 67,057 kDa, respectivamente. Presentan 79 residuos negativos y 70 residuos positivos, 280 residuos hidrofóbicos y 148 residuos hidrofílicos. Los perfiles de hidrofobicidad Kyte-Doolittle, Hoop-Woods y Eisenberg sugieren que el comportamiento general es hidrofóbico. Se determinaron 37 sitios potenciales de fosforilación, un sitio potencial de N-glicosilación y ausencia de sitios potenciales de O-glicosilación (figura 4).
Figura 4. Aminotrasferasa fructosa-6-fosfato glucosamina de H. Pylori J99.
- Perfiles de hidrofobicidad,
- Sitios potenciales de fosforilación.
- Sitios potenciales de N-glicosilación.
- Ausencia de sitios potenciales de O-glicosilación
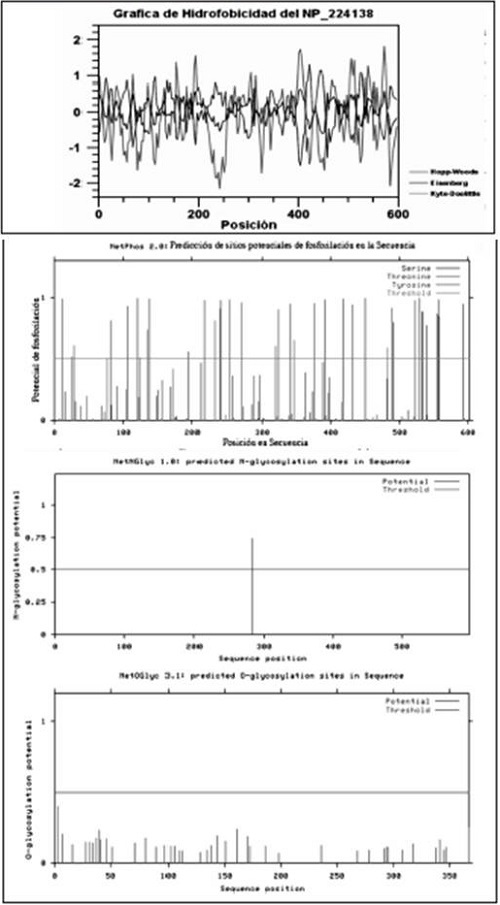
Fuente: elaboración propia.
3.1.3. Adhesina H. Pylori J99
Las adhesinas de H. Pylori son las responsables de su unión a receptores específicos de la mucosa del hospedero. Estas proteínas específicas han sido estudio de modelos teóricos para la fabricación de vacunas efectivas contra la infección por H. Pylori [21]. La secuencia de aminoácidos puede ser consultada en Gen- Bank con la referencia AAD06409 y en NCBI con la referencia NP_223551. En la figura 5 se aprecia la secuencia aminoacídica.
Figura 5. Adhesina de H. Pylori J99.
- Secuencia aminoacídica.
- Histograma de la distribución de
aminoácidos
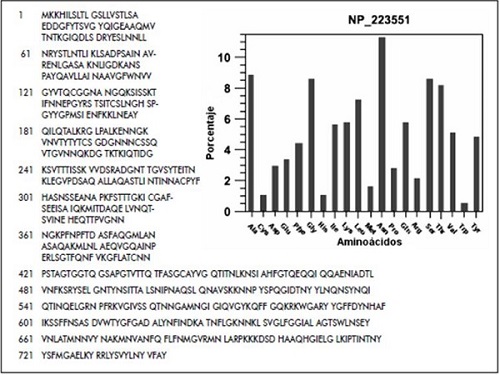
Fuente: elaboración propia.
Los valores de predicción computacional del punto isoeléctrico y el peso molecular de la secuencia son 9,06 y 80,6 kDa, respectivamente. En la proteína los residuos con carga negativa son 47 y con carga positiva, 59.
La cantidad de residuos hidrofóbicos que presenta la secuencia es de 334 y de residuos hidrofílicos es de 296. Los perfiles de hidrofobicidad Kyte-Doolittle, Hoop-Woods y Eisenberg muestran que los extremos de la cadena (tanto N-Terminal, como C-Terminal) son hidrofílicos y en el medio su comportamiento es hidrofóbico, lo que permite predecir que esta proteína es de transmembrana. En el análisis se determinaron 38 sitios potenciales de fosforilación, 6 sitios potenciales de N-glicosilación y ausencia de sitios potenciales de O-glicosilación (figura 6).
Figura 6. Adhesina de H. Pylori J99.
- Perfiles de hidrofobicidad,
- Sitios potenciales de fosforilación.
- Sitios potenciales de N-glicosilación.
- Ausencia de sitios potenciales de O-glicosilación
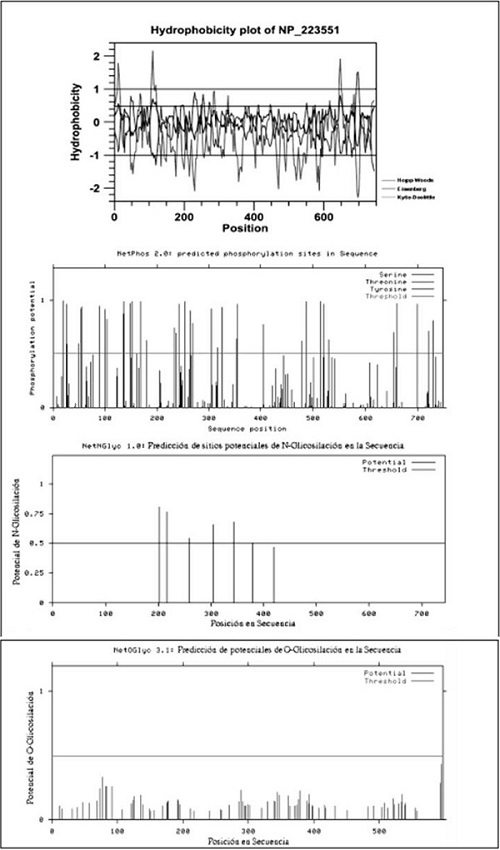
Fuente: elaboración propia.
La cantidad de residuos hidrofóbicos que presenta la secuencia es de 334 y de residuos hidrofílicos es de 296. Los perfiles de hidrofobicidad Kyte-Doolittle, Hoop-Woods y Eisenberg muestran que los extremos de la cadena (tanto N-Terminal, como C-Terminal) son hidrofílicos y en el medio su comportamiento es hidrofóbico, lo que permite predecir que esta proteína es de transmembrana. En el análisis se determinaron 38 sitios potenciales de fosforilación, 6 sitios potenciales de N-glicosilación y ausencia de sitios potenciales de O-glicosilación (figura 6).
3.1.4. UDP-N-acetilglucosamina1-car boxivinil-transferasa H. Pylori J99
La proteína UDP-N-acetilglucosamina1-carboxivinil- transferasa de H. Pylori J99 actúa como un importante activador de otras sustancias y transportador de fosfatos. La N-acetilglucosamina tiende a formar polisacáridos, principalmente de carácter estructural que cumplen varias funciones biológicas importantes en diversos procesos metabólicos. La unión de UDP y N-acetiglucosamina trabajan como un sustrato para diferentes enzimas (generalmente transferasas), hecho que favorece la glicosilación-fosforilación. La secuencia de aminoácidos para UDP-Nacetilglucosamina 1-carboxivinil transferasa de de H. Pylori J99 puede ser consultada en GenBank con la referencia AAD06166 y en NCBI con la referencia NP_223311. La secuencia aminoacidica se aprecia en la figura 7.
Figura 7. Transferasa 1-carboxivinil UDP-N-acetilglucosamina de H. Pylori J99.
- Secuencia aminoacídica.
- Histograma de la distribución de
aminoácidos
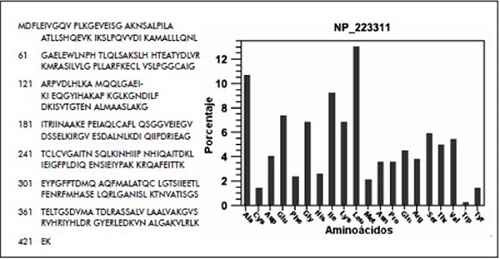
Fuente: elaboración propia.
Los valores de predicción computacional del punto isoeléctrico y el peso molecular de la secuencia son 6,69 y 45,81 kDa, respectivamente. La proteína presenta 48 residuos de carga negativa y 45 de carga positiva. Se encontraron 226 residuos hidrofóbicos y 95 residuos hidrofílicos. Los perfiles de hidrofobicidad Kyte-Doolittle, Hoop-Woods y Eisenberg muestran que la proteína posee tanto el dominio hidrofóbico, como el dominio hidrofílico, ya que el extremo N-Terminal es hidrofílico y el extremo C-Terminal es hidrofóbico. Se determinaron 12 sitios potenciales de fosforilación, un sitio potencial de N-glicosilación visto y ausencia de sitios potenciales de O-glicosilación (figura 8).
Figura 8. Transferasa 1-carboxivinil UDP-N-acetilglucosamina de H. Pylori J99.
- Perfiles de hidrofobicidad,
- Sitios potenciales de fosforilación.
- Sitios potenciales de N-glicosilación.
- Ausencia de sitios potenciales de O-glicosilación

Fuente: elaboración propia.
3.2. Análisis computacional de la estructura primaria de proteínas de H. Pylori 26695
La cantidad de proteínas codificadas para H. Pylori J99 es de 1523. Al igual que con el genoma anterior, se escogieron para un análisis bioinformático las siguientes proteínas: lipopolisacárido heptosiltransferasa-1, glucosamina- fructosa-6-fosfato-aminotransferasa, UDP-N-acetilglucosamina carboxiviltransferasa y adhesina flagelar, ya que estas están implicadas en la adhesión al epitelio gástrico y el metabolismo.
3.2.1. Lipopolisacárido heptosiltransferasa-1 de H. Pylori 26695
La secuencia de aminoácidos para lipopolisacárido heptosiltransferasa-1 de H. Pylori 26695 puede ser consultada en GenBank con la referencia AAD07342 y en NCBI con la referencia NP_207077. Los valores de predicción computacional del punto isoeléctrico y el peso molecular de la secuencia son 9,53 y 38,778 kDa, respectivamente. La secuencia presenta 35 residuos con carga negativa y 47 con carga positiva, 170 residuos hidrofóbicos y 80 residuos hidrofílicos. Los perfiles de hidrofobicidad Kyte-Doolittle, Hoop-Woods y Eisenberg sugieren que presenta una típica respuesta de una proteína transmembrana, la cual posee los dominios hidrofílicos e hidrofóbicos, el extremo N-Terminal es hidrofílico y el extremo CTerminal es hidrofóbico. Se determinaron 12 sitios potenciales de fosforilación, un sitio potencial de N-glicosilación y ausencia de sitios potenciales de O-glicosilación.
Figura 9. Lipopolisacarido heptosiltransferasa-1 de H. Pylori 26695. A.
- Secuencia aminoacídica.
- Histograma de la distribución de
aminoácidos
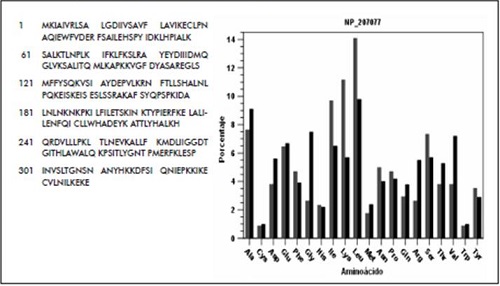
Fuente: elaboración propia.
Figura 10. Lipopolisacarido heptosyltransferasa-1 de H. Pylori 26695.
- Perfiles de hidrofobicidad,
- Sitios potenciales de fosforilación.
- Sitios potenciales de N-glicosilación.
- Ausencia de sitios potenciales de O-glicosilación
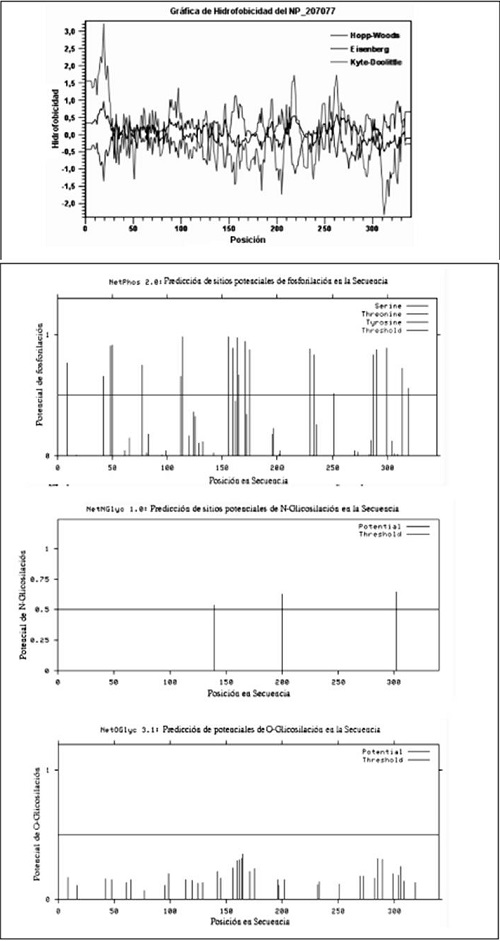
Fuente: elaboración propia.
3.2.2. Glucosamina-fructosa-6-fosfatoaminotransferasa H. Pylori 26695
La secuencia de aminoácidos para Glucosamina- fructosa-6-fosfato-aminotransferasa de H. Pylori 26695 puede ser consultada en Gen- Bank con la referencia AAD08570 y en NCBI con la referencia NP_208322. Los valores de predicción computacional del punto isoeléctrico y el peso molecular de la secuencia son 6,32 y 67,07 kDa, respectivamente. La cantidad de residuos hidrofóbicos que presenta la secuencia es de 279 y de 146 residuos hidrofílicos.
Los perfiles de hidrofobicidad Kyte-Doolittle, Hoop-Woods y Eisenberg sugieren que el comportamiento general tiende a ser hidrofóbico. Sin embargo, al analizar con detenimiento el extremo N-terminal se puede ver que es hidrofílico, lo que hace pensar en un dominio doble de la proteína. Se determinaron 37 sitios potenciales de fosforilación, un sitio potencial de N-y ausencia de sitios potenciales de O-glicosilación.
Figura 11. Glucosamina-fructosa-6-fosfato-aminotransferasa de H. Pylori 26695.
- Secuencia aminoacídica.
- Histograma de la distribución de
aminoácidos
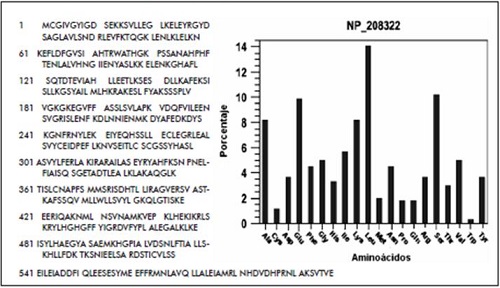
Fuente: elaboración propia.
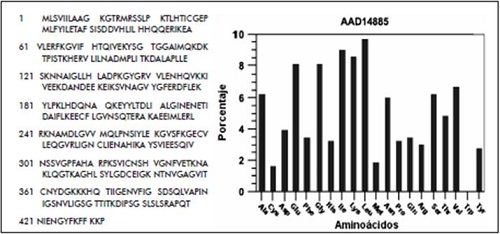
3.2.3. UDP-N-acetilglucosamina fosforilasa de H. Pylori 26695En el análisis de las proteínas de H. Pylori J99 se escogió el UDP-N-acetilglucosamina carboxiviltransferasa. Para este análisis se ha propuesto cambiar la enzima por fosforilasa para ver otro comportamiento metabólico en la bacteria. La fosforilasa es una enzima que degrada el glucógeno. La secuencia de aminoácidos para UDP-N-acetilglucosamina fosforilasa de H. Pylori 26695 puede ser consultada en GenBank con la referencia AAD14885 y en NCBI con la referencia NP_207477.
Figura 12. Glucosamina-fructosa-6-fosfato-aminotransferasa de H. Pylori 26695.
- Perfiles de hidrofobicidad,
- Sitios potenciales de fosforilación.
- Sitios potenciales de N-glicosilación.
- Ausencia de sitios potenciales de O-glicosilación
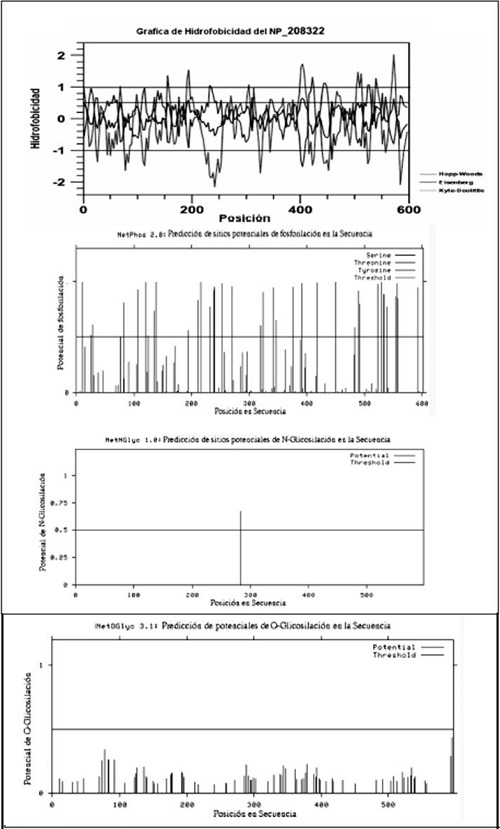
Fuente: elaboración propia.
punto isoeléctrico y el peso molecular de la secuencia son 7,01 y 47,802 kDa, respectivamente. La proteína presenta 52 residuos de carga negativa y 50 de carga positiva.
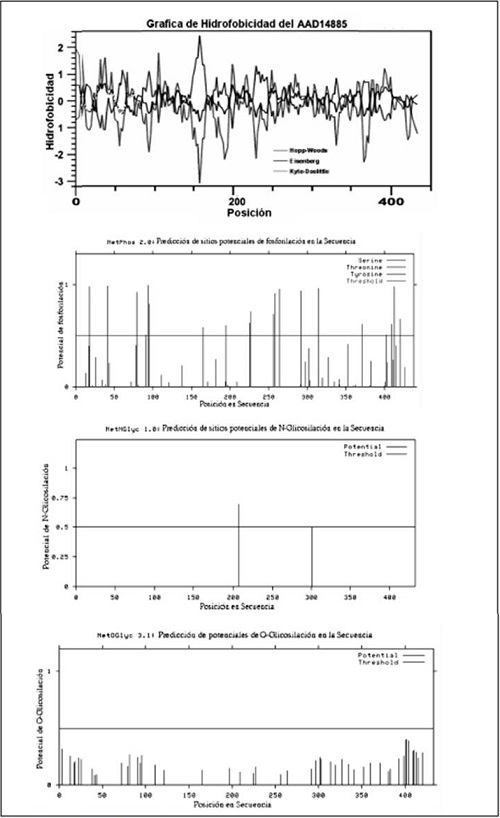
El análisis de hidrofobicidad determinó 209 residuos hidrofóbicos y 108 residuos hidrofilicos. Los perfiles de hidrofobicidad Kyte-Doo little, Hoop-Woods y Eisenberg reflejan un comportamiento hidrofóbico en general, aunque su extremo N-terminal es hidrofílico, así que posiblemente es una proteína con doble dominio. En los análisis también se determinaron veinte sitios potenciales de fosforilación, un sitio potencial de N-glicosilación y ausencia de sitios potenciales de O-glicosilación.
Figura 13. UDP-N-acetilglucosamina fosforilasa de H. Pylori 26695.
- Secuencia aminoacídica.
- Histograma de la distribución de aminoácidos en UDP-N-acetilglucosamina fosforilasa de
H. Pylori 26695
Fuente: elaboración propia.
Figura 14. UDP-N-acetilglucosamina fosforilasa H. Pylori 26695.- Perfiles de hidrofobicidad,
- Sitios potenciales de fosforilación.
- Sitios potenciales de N-glicosilación.
- Ausencia de sitios potenciales de O-glicosilación
Fuente: elaboración propia.
3.2.4. Adhesina flagelar de H. Pylori 26695
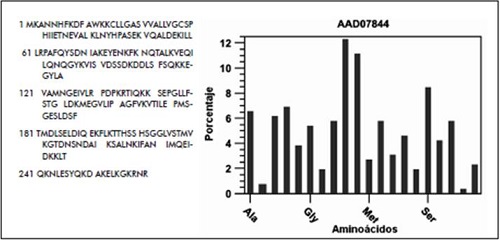
En el análisis al genoma de H. Pylori J99 se trabajó con una adhesina de membrana; sin embargo, otra estructura que está asociada al proceso de adhesión es la de los flagelos, que además de ser la principal estructura proveedora de movimiento de la bacteria, está recubierta de una proteína llamada adhesina flagelar que, junto con otras proteínas, forma la vaina o cobertura flagelar. Es a esta parte del flagelo a la que se le ha establecido un rol en la adherencia y protección del filamento flagelar; por eso la importancia de analizar esta proteína. La secuencia de aminoácidos para la adhesina flagelar de H. Pylori 26695 puede ser consultada en GenBank con la referencia AAD07844 y en NCBI CON NP_207077.
Figura 15. Adhesina flagelar de H. Pylori 26695.
- Secuencia aminoacídica.
- Histograma de la distribución de aminoácidos
Fuente: elaboración propia.
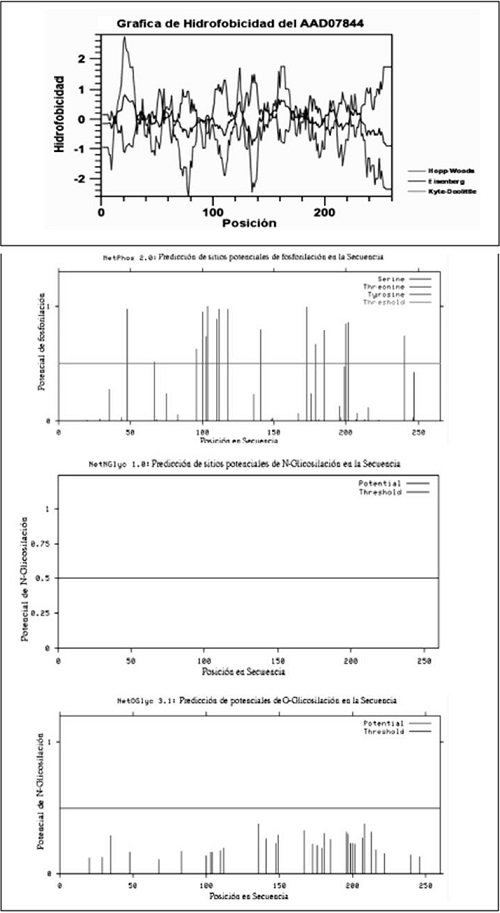
Los valores de predicción computacional del punto isoeléctrico y el peso molecular de la secuencia son 8,96 y 29,04 kDa, respectivamente. Los análisis muestran que la proteína presenta 34 residuos de carga negativa y 37 de carga positiva. Sobre el comportamiento de la carga de la proteína en función del pH del medio, sugieren que se comporta con carga positiva mientras el pH no supere su punto isoeléctrico, el cual tiene un valor de 9,03. Cuando se encuentra en un medio básico con pH superior a 9,03, la proteína tendrá un comportamiento de carga negativo.En la proteína están contenidos 116 residuos hidrofóbicos y 68 residuos hidrofílicos. Los perfiles de hidrofobicidad Kyte-Doolittle, Hoop-Woods y Eisenberg indican que el comportamiento de la proteína es hidrofóbico, aunque hay unos picos hidrofílicos entre los aminoácidos 5 y 30, así como entre los 140 y 160 aproximadamente. Se determinaron 12 sitios potenciales de fosforilación, un sitio potencial de N-glicosilación y ausencia de sitios potenciales de O-glicosilación.
Figura 16. Adhesina flagelar de H. Pylori 26695.
- Perfiles de hidrofobicidad,
- Sitios potenciales de fosforilación.
- Sitios potenciales de N-glicosilación.
- Ausencia de sitios potenciales de O-glicosilación
Fuente: elaboración propia.
A pesar de los avances en la descripción microbiológica, taxonómica y genética de H. Pylori [22], [23], es necesario analizar proteínicas capaces de inactivar esta bacteria. El H. Pylori es una bacteria gram-negativa con pared celular conformada por una secuencia alternante de N-acetilglucosamina y N-acetilmurámico, el ácido m-diaminopimélico y las formas D de la alanina y del ácido glutámico, que constituyen el conjunto llamado “red de mureína”, esqueleto de la pared celular [24]. Considerando la composición de la pared celular, se sugiere a las glucoproteínas como blancos de encapsulamiento, considerando que son proteínas que contienen cadenas de oligosacáridos y están unidas por enlaces covalentes a sus esqueletos polipetídicos. Sus funciones más destacadas son moléculas estructurales, molécula inmunológica, función enzimática e incluso son sitios de reconocimiento para la adhesión celular [25], [26]; es necesario, además, determinar el tipo de glucoproteína. Inicialmente se pueden clasificar en glucoproteínas de enlace O-glicosídico, glucoproteínas de enlace N-glicosídico e inmunoglobulinas. En el análisis de sitios potenciales de fosforilación se encontraron sitios potenciales en cada proteína analizada, y se confirmó así la complejidad de su señalización y comunicación celular. Este hecho complica el estudio de antígenos monoclonales que permitan atacar la bacteria e inhibir el daño hístico que produce; así mismo, como la función esperada es el encapsulamiento, las inmunoglobulinas no tendrían la capacidad de realizarla [27], [28]. A todas las proteínas analizadas se les realizó un estudio de sitios potenciales de N-glicosilación y O-glicosilación. Aunque los resultados mostraron que los sitios de N-glicosilación son pocos (entre 1 y 6), por lo menos la mayoría de proteínas analizadas tiene sitios potenciales. En cambio, hay ausencia total de sitios potenciales de O-glicosilación.
El enlace N-glicosídico se da a través de la vinculación al grupo amida de asparagina. El carbohidrato que se adhiere directamente a la proteína es N-acetilglucosamina (GlcNAc) [25]. Las N-glucoproteínas se encuentran vinculadas dentro de un consenso secuencia de aminoácidos, N-X-S/T, donde X es cualquier aminoácido excepto prolina. Cuando un análisis de las proteínas en las bases de datos públicas se lleva a cabo, se puede demostrar que aproximadamente el 65 % de todas las proteínas contienen al menos una ocurrencia de la Asn-X-Ser/Thr consenso [21], [25]. Por medio de esta información se logró predecir que la familia de las glucoproteínas N-glicosilisadas específicamente con radicales de Nacetilglucosamina pueden llegar a encapsular la bacteria, ya que si se lograra combinar una glucoproteína con N-acetilglucosamina como residuo y que esta se una al esqueleto de la pared celular de la bacteria, se podría construir una membrana que encapsule la bacteria. Recientemente, Masatomo Kawakubo y sus colaboradores encontraron que incluso la mucosa gástrica posee un antibiótico natural capaz de inhibir al H. Pylori: la glucoproteína alfa 1,4 N-acetilglucosamina [29].
4. Conclusiones
Los puntos isoeléctricos de las proteínas estudiadas se encuentran alrededor de 6,32 y 9,53 y corresponden a proteínas transmembrana, exceptuando las adhesinas. Se encontraron entre 10 y 38 sitios potenciales de fosforilación, entre 1 y 6 sitios potenciales de N-glicosilación y ausencia de sitios potenciales de O-glicosilación.
Referencias
- R. Murillo, P. Pineros y G. Hernández Suarez, Atlas de mortalidad por cáncer en Colombia, Instituto Nacional de Cancerología e Instituto Geográfico Agustín Codazzi. Imprenta Nacional de Colombia, 2003.
- R. Puffer y G. W. Griffith. “Cáncer,” en Características de la mortalidad urbana. Organización Panamericana de la Salud. Washington D. C.: Organización Mundial de la Salud, 1968.
- A. Giraldo, “Tasa de mortalidad por cánceres del tubo digestivo según género y grupo de edad en Colombia entre 1980 y 1998,” Revista Colombiana de Gastroenterología,,vol. 23, no. 2: 124- 135, abril / junio 2008. [En línea]. Disponible en http://www.gastrocol.com/ file/Revista/v23n2a05.pdf
- IARC, Monographs on the evaluation of carcinogenic risks to humans. Schistosomes, liver flukes and Helicobacter pylori. Vol. 61. Lyon: International Agency for Research on Cancer, 1994.
- M. Kidd y I. M. Modlin, A Century of Helicobacter Pylori: Paradigms Lost- Paradigms Regained. Digestion, vol. 59, no. 1, pp. 1-15. 1998.
- F. Rivas, F. Hernández. Helicobacter pylori: Factores de virulencia, patología y diagnóstico. Rev Biomed; 11:187-205, 2000.
- L. Bravo, “Helicobacter pylori: patología y prevalencia en biopsias gástricas en Colombia,” Colombia Médica, vol. 34, no. 3: 124-131, 2003. [En línea]. Disponible en http://redalyc. uaemex.mx/pdf/283/28334303.pdf
- C. Pelayo, y B. Piazuelo, Cáncer gástrico: el enigma colombiano. Asociaciones Colombianas de Gastroenterología, Endoscopia digestiva, Coloproctología y Hepatología, Bogotá, 2010.
- A. Alm, L Ling, D Moir, B King, et. al., “Genomic-sequence comparison of two unrelated isolates of the human gastric pathogen Helicobacter pylori,” Nature,
- J. Tomb, O. White, A. Kerlavage, R. Clayton, vol. 338, no. 6642 et al., “The complete genome sequence of the gastric pathogen Helicobacter pylori,”. Nature, vol. 6642, no. 388, pp. 539-547, 1997.
- W. R. Pearson, “Rapid and sensitive sequence comparison with FASTP and FASTA,” Methods Enzymol, vol. 183, pp. 63-98, 1990.
- J. Kyte y R. F. Doolittle, “A simple method for displaying the hydropathic character of a protien,,” Journal of Molecular Biology, vol. 157, no. 1, pp. 105-132, mayo 1982.
- T. P. Hoop y K. R. Woods, “Prediction of protein antigenic determinants from amino acid sequences,” Proc Natl Acad Sci, Vol. 78, no. 16, p. 3824, junio 1981.
- D. Eisenberg, E. Schwarz, M. Komarony, Wall R Amino acid scale: Normalized consensus hydrophobicity scale. J. Mol. Biol.179:125-142. 1984
- N. Blom, S. Gammeltoft y S. Brunak, “Sequence and structure-based prediction of eukaryotic protein phosphorylation sites,” Journal of Molecular Biology, vol. 294, no. 5, pp. 1351-1362, diciembre 1999.
- K. Julenius, G. Molgaard, R. Gupta y S. Brunak, “Prediction, conservation analysis and structural characterización of mammalian mucin-type O-glycosylation sites,” Glycobiology, vol. 15, pp. 153-164. 2005.
- N. Blon, T. Ponten, R. Gupta y S. Brunak S., “Prediction of post-translational glycosilation and phosphorylation of protein from aminoacid sequence,” Proteomics, vol. 4, pp. 1633-1649, 2004
- J. Piotrowski, E. Piotrowski, D. Skrodzka, et. al., “Induction of acute gastritis an epithelial apoptosis by Helicobacter Pylori lipopolysaccharide,” Scand Gastroenterol. Vol. 32, no. 3, pp. 203- 211, 1997.
- N. Rojas, El lipopolisacárido bacteriano: Una potente endotoxina con múltiples actividades biológicas. Costa Rica: Universidad de Costa Rica, 1995.
- A. Lichtman, et. al., Inmunología celular y molecular. Madrid: Interamericana McGraw-Hill, 2000.
- M. Nuñez, V. Scialom, D. Silva, et al., “Modelo teórico para una vacuna efectiva contra la infección por Helicobacter pylori,” Vitae, no. 28, 2006.
- A. Marais, GL. Mendz, S. Hazell, F. Mégraud. Metabolism and Genetics of Helicobacter pylori: the Genome Era. Microbiology and Molecular Biology Reviews, vol. 63, no. 3, pp. 642-674, 1999.
- R. A. Alm y B. Noonam, “The Genome,” en Helicobacter pylori: Physiology and Genetics. Washington, ASM Press, 2001.
- H. Schlegel, Microbiología general. Barcelona, Editorial Omega. 1997.
- R. K. Murray, P. A. Mayes, D. K. Granner y V. W. Rodwell, Bioquímica de Harper. México: Editorial Manual Moderno, 2001.
- A. Lehninger, Bioquímica, Barcelona, Editorial Omega, 1995.
- H. Kleanthous, G. A Myers y J. W. Ingrassia Rectal and intranasal immunizations with recombinant urease induce distinct local and serum immune responses in mice and protect against Helicobacter Pylori infection, Infection and Immunity, vol. 66, no. 6: 2879- 2886, junio 1998.
- O. G Gómez, B. Lucas, Z. X. Yan, K. Panthel, T. F.Haas R., “Meyer. Protection of mice against gastrict colonization by Helicobacter Pylori by single oral dose immunization with attenuated Salmonella typhimurium producing urease subunits A and B,” Vaccine vol. 16, no.5: 460-471, marzo 1998.
- M. Kawakubo, Y. Ito, M. Fukuda, et al., “Natural Antibiotic Function of a Human Gastric Mucin Against Helicobacter Pylori Infection,” Science, vol 305, no. 5686, pp. 1003-1006, agosto 2004.
Creation date: Diciembre de 2012


.png)




.jpg)