DOI:
https://doi.org/10.14483/23448393.12512Published:
2019-05-26Issue:
Vol. 24 No. 2 (2019): May - AugustSection:
Electrical, Electronic and Telecommunications EngineeringDetección de Candidatos a Microcalcificaciones Mamarias Agrupadas en Mamografías
Clustered Microcalcifications Candidates Detection in Mammograms
Downloads
References
Sociedad Americana Contra el Cáncer, ¿Que es el cáncer de seno?, [En línea], 2016. Disponible en: https://www.cancer.org/es/cancer/cancer-de-seno/acerca/que-es-el-cancer-de-seno.
R. Buffa, S. Gamarra, and M. Viniegra, “Manual operativo para el uso de mamografía en tamizaje,” Programa de Control de Cancer de mama. Cazap S, editor. Argentina, 2012.
Ministerio de Salud y Protección Social, Cáncer de mama, una enfermedad en ascenso en Colombia [En línea], 2014. Disponible en: https://www.minsalud.gov.co/Paginas/-Cancer-de-mama,-una-enfermedad-en-ascenso-en-Colombia.
S. Díaz, M. Piñeros, and O. Sánchez, “Detección temprana del cáncer de mama: aspectos críticos para un programa de tamizaje organizado en Colombia.,” Revista colombiana de cancerología, no. 528, pp. 93–105, 2005.
L. A. Villán, “Clasificación radiológica de las lesiones mamarias y manejo,” Servicio de Obstetricia y Ginecología Hospital Universitario Virgen de las Nieves, pp. 1–19.
K. Junemann, “Patología mamaria e imágenes,” Obstet ginecol Hosp Santiago Oriente Dr. Luis Tisné Brousse.[Internet], vol. 4, no. 3, pp. 189–194, 2009.
L. M. Mina and A. Mat, “Microcalcification Diagnosis in Digital Mammograms Based OnWavelet Analysis and Neural Networks,” 2015 IEEE International Conference on Control System, computing and Engineering, vol. 1, no. November, pp. 27–29, 2015. https://doi.org/10.1109/ICCSCE.2015.7482149
M. Mustra and M. Grgic, “Detection of Areas Containing Microcalcifications in Digital Mammograms,” in lWSSIP 2014, 21st International Conference on Systems, Signals and Image Processing, no. May, pp. 12–15, 2014.
A. Mencattini, G. Rabottino, M. Salmeri, and R. Lojacono, “A study on the automatic detection of clustered microcalcifications in mammograms,” 2010.
P. Arancibia, T. Taub, and K. De Grazia, “Microcalcificaciones mamarias: revisión de los descriptores y categorías BI-RADS,” Revista Chilena de Obstetricia y Ginecología., vol. 78, no. 5, pp. 383–394, 2013. https://doi.org/10.4067/S0717-75262013000500010
T. Balakumaran, I. Vennila, and C. Shankar, “Detection of Microcalcification in Mammograms Using Wavelet Transform and Fuzzy Shell Clustering,” International Journal of Computer Science and Information Security, vol. 7, no. 1, pp. 121–125, 2010.
A. Papadopoulos, D. I. Fotiadis, and L. Costaridou, “Improvement of microcalcification cluster detection in mammography utilizing image enhancement techniques,” Computers in biology and medicine, vol. 38, no. 10, pp. 1045–1055, 2008. https://doi.org/10.1016/j.compbiomed.2008.07.006
L. Vivona, D. Cascio, F. Fauci, and G. Raso, “Fuzzy technique for microcalcifications clustering in digital mammograms,” BMC medical imaging, vol. 14, no. 1, p. 23, 2014. https://doi.org/10.1186/1471-2342-14-23
E. Hashemi Aghjekandi, “Microcalcification detection in mammography using wavelet transform and statistical parameters,” 2012.
M. Mustra, M. Grgic, and K. Delac, “Enhacement of microcalcifications in digital mammograms,” IWSSIP 2012, no. April, pp. 11–13, 2012.
A. Oliver, A. Torrent, X. Lladó, M. Tortajada, L. Tortajada, M. Sentís, J. Freixenet, and R. Zwiggelaar, “Automatic microcalcification and cluster detection for digital and digitised mammograms,” Knowledge-Based Systems, vol. 28, pp. 68–75, 2012. https://doi.org/10.1016/j.knosys.2011.11.021
A. Khalaf and I. Yassine, “Novel features for microcalcification detection in digital mammogram images based on wavelet and statistical analysis,” in ICIP, pp. 1825–1829, 2015. https://doi.org/10.1109/ICIP.2015.7351116
P. J. García Laencina et al., “Mejora en la detección de microcalcificaciones en mamografías digitalizadas mediante la aplicación de arquitecturas neuronales,” 2008.
S. Mallat, “A Theory for Multiresolution Signal Decomposition : The Wavelet Representation,” IEEE Transactions on Pattern Analysis and Machine Intelligence, vol. I, no. 7, pp. 674–693, 1989. https://doi.org/10.1109/34.192463
How to Cite
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Download Citation
Recibido: 3 de agosto de 2018; Revisión recibida: 27 de febrero de 2019; Aceptado: 10 de mayo de 2019
Resumen
Contexto:
Las microcalcificaciones mamarias son lesiones no palpables que están presentes aproximadamente en el 55% de los casos de cáncer de mama; son un hallazgo muy frecuente en mamografías y pueden ser un indicador de esta enfermedad en sus primeras fases.
Método:
Se implementó un método para el realce de microcalcificaciones mamarias basado en análisis multirresolución a través de la transformada wavelet. Posteriormente se realiza una segementación aproximada de candidatos utilizando los parámetros estadísticos de la distribución de coeficientes y se seleccionaron aquellos que cumplían con ciertos parámetros de longitud y distribución. Por último, se se emplea un modelo de clasifiación binaria utilizando maquinas de soporte vectorial para clasificar imagenes con microcalcificaciones mamarias agrupadas.
Resultados:
Se realizó la tarea de clasificación binaria entre imágenes con tejido mamario sano y con microcalcificaciones utilizando una máquina de soporte vectorial (Support-vector machine, SVM, por sus siglas en inglés) utilizando kernel lineal. Las métricas de evaluación para el algorítmo fueron: área bajo la curva (AUC) de 93.6 %, precisión de 89.4 %, sensibilidad de 88.4% y especificidad de 90.5 %.
Conclusiones:
En el presente trabajo se utilizó el tamaño y la distribución como criterios para la segmentación y caracterización de microcalcificaciones mamarias agrupadas, que en la evaluación clínica son medidas cuantitativas para la detección de cáncer de seno en sus primeras etapas. Dado que el tejido mamario presenta un bajo contraste se propuso un método de realce que logra desenmascarar algunas microcalcificaciones que son poco evidentes. Con el enfoque propuesto se logró clasificar correctamente la mayoría de imágenes de tejido mamario con microcalcificaciones agrupadas ya sea en distribuciones segmentarias, lineales o ramificadas.
Palabras clave:
análisis multirresolución, detección de cáncer de seno, mamografía, microcalcificaciones mamarias, transformada Wavelet, Idioma: Español.Abstract
Context:
Mammary microcalcifications are not-palpable lesions that are present in approximately 55% of breast cancer. These are a frequent findings in mammograms and may be an indicator of the disease in its early stages.
Method:
A method was implemented in order to get mammary microcalcifications enhancement based on multi-resolution analysis with Wavelet transform. Then, candidates were segmented using thresholding, in this technique, the threshold was determined with statistical parameters from Wavelet distribution coefficients. Later, a couple of Support Vector Machines models was used to classify images that contains mammary microcalcifications.
Results:
Classification task was performed using Support Vector Machines (SVM). The following evaluation metrics was achieved: AUC of 93.6 %, accuracy of 89.4 %, sensivity of 88.4%and specificity of 90.5%
Conclusions:
In this approach the length and distribution of microcalcifications was used as features to select candidates. These features are also used as criteria in clinical evaluation to detect mammary cancer in early stages. The proposed method to image enhancement can unmask microcalcifications that are not visible at naked eye. In most mammographies the proposed algorithm classify correctly microcalcifications in different distributions.
Keywords:
Breast cancer detection, mammary microcalcifications, mammography, multi-resolution analysis, wavelet transform, Language: Spanish.1. Introducción
El cáncer de seno se origina por el crecimiento descontrolado de células, el cual se puede localizar en diferentes partes y puede afectar los lóbulos mamarios (carcinoma lobular) o en su mayoría a los conductos lácteos (carcinoma ductual) [1]. Es considerado como la segunda causa de muerte por cáncer en mujeres en el mundo [2]. En Colombia, es el cáncer más común. Se estima que anualmente fallecen más de 2000 mujeres por esta causa y se reportan más de 7 mil nuevos casos [3].Por este motivo, se ha convertido en un problema de gran magnitud en la población femenina que requiere la implementación de estrategias para la prevención, detección temprana, control y tratamiento adecuado de la enfermedad [4].
La mamografía o mamograma es una imagen radiológica del seno que sirve para detectar cáncer en sus primeras etapas. Es el único método de diagnóstico que ha demostrado una reducción cercana al 30% en las tasas de mortalidad por cáncer [5] [6].
Los hallazgos más comunes en las mamografías son las microcalcificaciones, lesiones no palpables en la mama que se caracterizan por ser pequeños depósitos de calcio. Algunas características como el patrón de distribución dentro de la mama y la morfología indican la formación de alguna lesión maligna [7]; por ejemplo, las microcalcificaciones que tienen una longitud menor a 0.5 mm y que en un área menor o igual a 1 cm2 de tejido se agrupan más de tres, indican un alto grado de sospecha de malignidad [8] [9].
En el tejido mamariola detección de microcalcificaciones ha sido tema de interés para los investigadores,ya que dichos elementos están presentes aproximadamente en el 55% de los casos de cáncer de mama no palpable [10]. Además supone un reto, ya que las microcalcificaciones son tan pequeñas que a simple vista entre el 10% y el 40% pasan desapercibidas [11].
La interpretación de mamografías es una tarea que requiere tiempo y esfuerzo, además de la experiencia y la habilidad del médico especialista. El diagnóstico asistido por computador (Computer Aided Diagnosis, CAD, por sus siglas en inglés) sirve como soporte para esta labor. Por ejemplo:mediante el mejoramiento de la calidad de la imagen, el realce de regiones sospechosas, y la identificación de elementos de interés.
En la literatura se han propuesto algunas estrategias para la detección de microcalcificaciones en imágenes mamográficas Papadopoulos et al. [12] presentan cinco algoritmos de mejora de imagen y el ajuste de parámetros para la detección de los píxeles con mayor contraste y de tamaño mínimo que pueda representar satisfactoriamente una microcalcificación, dando como resultado un área bajo la curva de 93 %. Por su parte Vivona et al. [13] propone un algorítmo de agrupamiento difuso basado en C-Means (FCM) para la detección de agrupaciones de microcalcificaciones conociendo previamente la ubicación del grupo dentro del seno y el número exacto de microcalcificaciones.Este método fue evaluado en términos de sensibilidad (93 %) y precisión (95 %). Hashemi [14] plantea el uso de análisis estadístico de orden superior (High Order Statistics, HOS, por sus siglas en inglés) en los coeficientes Wavelet, en donde los puntos de cruce entre filas y columnas asociados a un alto sesgo y curtosis determinan las regiones con agrupaciones de microcalcificaciones.Por otro lado, Mustra et al. [15] elaboran un método para mejorar el contraste de las imágenes mamográficas y resaltar las microcalcificaciones suprimiendo el fondo de la imagen, y lo logran a través de la combinación entre el análisis multirresolución y morfología en escala de grises [8]. Por su parte Oliver et al. [16] propone el uso de características locales extraídas de bancos de filtros para obtener una descripción local de la morfología de las microcalcificaciones. El desarrollado del enfoque realiza un paso de entrenamiento inicial a fin de seleccionar automáticamente las funciones más destacadas y que posteriormente se utilizan en un clasificador para realizar la detección de microcalcificaciones individuales. Sin embargo, amplían el método para la detección de microcalcificación agrupadas logrando un área bajo la curva de 91.8 %. Khalaf et al. [17] presenta un enfoque basado en descomposiciónWavelet y análisis de parámetros estadísticos de orden superior (HOS) que junto a la prueba t-Student evalúa y reduce las características; en la clasificación utiliza máquinas de soporte vectorial (Support Vector Machines, SVM, por sus siglas en inglés) con kernel RBF, logrando una sensibilidad de 96.7 %, especificidad de 93.3% y una precisión de 95.8 %.
En este trabajo se propone un método para el realce basado en la eliminación sub-banda utilizando la transformada Wavelet y la posterior segmentación de candidatos a microcalcificaciones agrupadas en términos de su longitud y distribución. Dado que se utiliza una base de datos validada,se extraen imagenes cuadradas con tejido sano y con presencia de microcalcificaciones mamarias y se caracterizan a fin de entrenar un modelo de clasificación binaria utilizando máquinas de soporte vectorial (SVM).
2. Metodología
En la primera parte de esta sección, se describe el procedimiento para el realce y la segmentación de candidatos a microcalcificaciones agrupadas en imágenes mamográficas y la caracterización de los parches cuadrados de tejido mamario. En la parte final se construye una matriz de características para entrenar un modelo de clasificación binaria entre parches de tejido sano y con microcalcificaciones utilizando máquinas de soporte vectorial (SVM).
2.1. Captura
Se emplearon 86 imágenes mamográficas de 43 casos que dispone la Universidad del Sur de la Florida en su base de datos pública DDSM (Digital Database for Screening Mammography,DDSM, por sus siglas en inglés). En cada caso se cuenta con cuatro imágenes que corresponden a las proyecciones medio lateral oblicua (MLO) y cráneo caudal (CC) para cada seno [18]. Todas las imágenes tienen una resolución espacial en un rango entre 50 a 80 _m por píxel con una profundidad de 12 bits.
2.2. Extracción de regiones cuadradas
Para este trabajo se cuenta con una base de datos validada, y en consecuencia se conoce la localización y el contorno de las microcalcificaciones agrupadas en distibuciones segmentarias, lineales y ramificadas. En este sentido, se extraen regiones o parches cuadrados de 200 pixeles de lado como se muestra en la Figura 1.
Figura 1: a) Imagen mamográfica original. b) Parche con microcalcificaciones. c) Parche de tejido sano.
2.3. Caracterización de microcalcificaciones
Las microcalcificaciones en las imágenes mamográficas tienen la cualidad de ser agrupaciones de píxeles relativamente brillantes comparados con sus píxeles adyacentess. Con motivo de las bajas dosis y corto tiempo de exposición a la radiación a la que son sometidas las pacientes, las imágenes presentan bajo contraste, causando que entre el 10 y el 40% de estas lesiones pasen desapercibidas por el experto [11]. Por esta razón, el realce de estos elementos en la imagen es un paso significativo para la posterior segmentación [8].
2.3.1. Realce de candidatos a microcalcificaciones
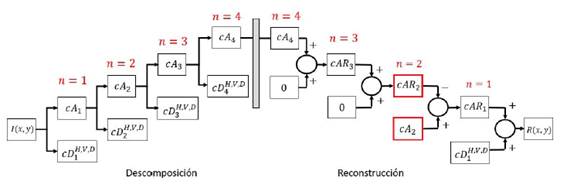
En método de realce de microcallcificaciones propuesto se emplea análisis multirresolución (Multiresolution Analysis, MRA, por sus siglas en inglés) y supresión sub-banda a fin de reducir la densidad del tejido mamario y realzar los detalles finos y brillantes de las mamografías. Inicialmente se descompone la región cuadrada o parche de tejido mamario mediante la transformada discreta Wavelet (Discrete wavelet transform, DWT, por sus siglas en inglés) que a través de un banco de filtros ortogonales pasa bajo y pasa alto se obtiene la representación de la imagen inicial en cuatro matrices de coeficientes wavelet o sub-bandas: los coeficientes de aproximación y los coeficientes de detalles horizontales, verticales y diagonales respectivamente [7), [11], [19]. Cabe destacar que en el análisis multirresolución, por cada nivel n de descomposición el número de filas y columnas de las matrices se reducen, por consiguiente la resolución de la imagen original decrece en un factor de 2n [15]. Por ese motivo, el parche cuadrado solo se descompone hasta el cuarto nivel de resolución dado que allí los coeficientes de aproximación muestran una representación tosca del tejido mamario. Posteriormente, se reconstruye la imagen a través de la transformada discreta wavelet inversa (IDWT) (Inverse Discrete Wavelet Transform, IDWT, por sus siglas en inglés) y se suprimen las sub-bandas correspondientes a los coeficientes de detalle horizontales, verticales y diagonales desde el cuarto hasta el segundo nivel de reconstrucción, lo que reduce las altas frecuencias,logrando una representación gruesa y sin detalles finos del tejido mamario en términos de los coeficientes de aproximación en el segundo nivel como se muestra en el esquema de la Figura 2.
Figura 2: Esquema de descomposición y reconstrucción de la imagen para el realce de micro calcificaciones. Donde cAn son los coeficientes de aproximación, cDH;V;D n ; son los coeficientes de detalle horizontal, vertical y diagonal del nivel de descomposición y reconstrucción
Ahora bien, dado que en el segundo nivel la matriz de coeficientesWavelet de aproximación en la etapa de descomposición presenta informacion aproximada de la imagen original y, por otro lado,la matriz de coeficientes de aproximación en fase de reconstrucción en el mismo nivel muestra una representación gruesa y sin detales del tejido mamario se procede a realizar una sustracción entre ambas matrices de coeficientes dado que tienen el mismo tamaño, dando como resultado una representación que resalta las microcalcificaciones y otros elementos brillantes y pequeños como se aprecia en la Figura 3; por último, se transforma la imagen hasta su tamaño original.
Figura 3: a) Imagen original b) Realce de microcalcificaciones c) Imagen umbralizada d) Elementos segmentados por Longitud.
2.3.2. Segmentación de candidatos a microcalcificaciones
Una vez se tiene la matriz de coeficientes que resulta del método de realce descrito anteriormente se explora la distribución de dichos elementoss, notando que se distribuyen aproximadamente de forma normal o gaussiana. Por lo tanto, se propone segmentar los candidatos a microcalcificaciones partiendo de las siguientes hipotesis: La primera sugiere que las microcalcificaciones pueden ser segmentadas utilizando un umbral cercano a los coeficientes cuyo valor en magnitud sea alto; lo cual conduce a la segunda hipótesis, que se basa en la baja presencia de microcalcificaciones en las imágenes, por consiguiente se plantea que la probabilidad de que un píxel sea parte de una microcalcificación es baja; dado lo anterior, se puede relacionar el histograma de coeficientes con una función de densidad de probabilidad normal. Por lo tanto, se plantea la umbralización para la segmentación de candidatos a microcalcificaciones con la siguiente expresión:
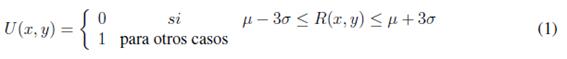
donde U(x; y) es una imagen binaria, R(x; y) es la imagen realzada, μ es el valor medio y σ es la desviación estándar de la distribución de niveles de gris. La segmentación utilizando este umbral se puede ver en la Figura 3 c.
2.3.3. Selección a partir de la longitud
Una vez que se logra la segmentación de los candidatos a microcalcificaciones, algunos otros elementos son también segmentados causando un aumento considerable de falsos positivos. Para reducir esto, se propone clasificar los elementos a partir de su longitud, considerando que una microcalcificación es sospechosamente maligna cuando tiene una longitud menor a 1 mm [8] se descartan todos aquellos elementos que estén por fuera de este límite, como se evidencia en la figura 3 d. Cabe aclarar que se tiene en cuenta la relación espacial de la imagen y su equivalente píxeles, que para esta investigación se utilizaron imágenes con una resolución espacial entre 50 y 80μm/pix [11] y en consecuencia, la longitud de los candidatos a microcalcificaciones en pixeles se calcula con la siguiente expresión:

donde Lpix es la longitud en pixeles, Lm es la longitud en μm de las microcalcificaciones y Re es la resolución espacial de la imagen. Por ejemplo, para una microcalcificación de 1 mm de longitud en una imagen de resolución espacial de 50μm/pix se tiene que:

2.3.4. Selección a partir de la distribución
Para esta etapa se tiene en cuenta que las microcalficaciones se consideran de alta sospecha de malignidad cuando tres o más se agrupan en un área menor a 1cm2. Sobre la base de ello se decide seleccionar los candidatos utilizando el algoritmo de agrupamiento basado en densidad (Density Based Clustering, DBC, por sus siglas en inglés). Este algoritmo consiste en conectar los objetos de la imagen binaria mediante un umbral de distancia, con el propósito de definir las áreas con más alta densidad dentro de la imagen. El procedimiento consiste calcular la distancia euclidiana entre los objetos y se organiza en una matriz de distancias tal como se muestra en la ecuación (4):
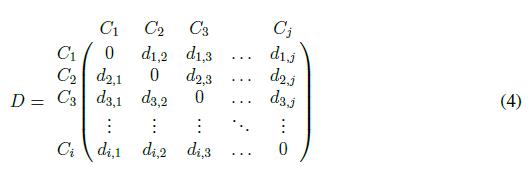
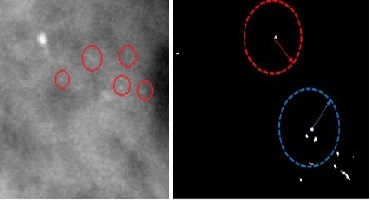
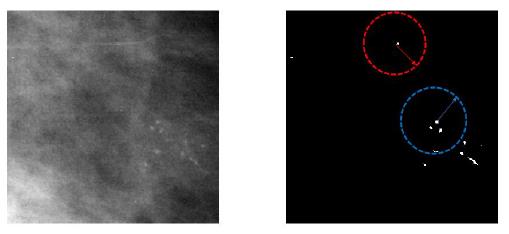
donde los elementos di;j de la matriz corresponden a la distancia euclidiana entre los centroides de los objetos Ci y Cj de la imagen binaria (Figura 4).
Figura 4: La imagen de la izquierda muestra el tejido mamario con una agrupación de microcalcificaciones. La imagen de la derecha muestra la segmentación de los candidatos a microcalcificaciones. El círculo de color azul corresponde a la región radial de un candidato a microcalcificación con 3 vecinos. El circulo color rojo corresponde a la región de un elemento que se descarta como candidato a microcalcificación por no tener vecinos dentro de la región radial
A partir de la matriz D de la expresión 4 se genera una nueva matriz D*, en donde los elementos di;j son reemplazados por cero cuando superan el umbral de distancia RTh, o en caso contrario tomarán el valor de uno, la expresión que realiza la desagregación es:
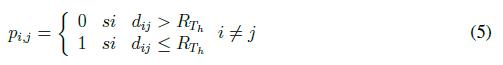
Lo cual lleva a que la matriz D* quede reducida de la siguiente forma:
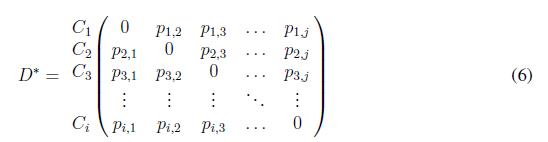
Cuando la suma de los elementos de cada fila de la matriz D_ es mayor o igual a tres, el objeto Cise considera candidato a microcalcificación agrupada como se muestra en la siguiente exrpresión:

donde Fi es la sumatoria de los elementos pi;j de la fila i de la matriz D*.
Este método visto de otra manera, consiste en fijar una distancia radial (RTh) que para este caso es de 0,5 cm o 20 píxeles desde el centroide de cada objeto Ci y se determina el número de vecinos dentro de la circunferencia trazada con dicho radio. Si cumple con la condición de tener 3 o más vecinos, será clasificado como candidato a microcalcificación agrupada, pero en el caso contrario,el elemento será descartado como se muestra en la Figura 4.
2.4. Clasificación
Las máquinas de soporte vectorial (SVM) corresponden a métodos de aprendizaje supervisados que comunmente se relacionan con problemas de clasificación y regresión. El clasificador establece un hiperplano que transforma el espacio de entrada mediante un kernel a un espacio de características y busca la máxima distancia que separa las dos clases. El modelo de clasificación fue entrenado con una matriz de características construida a partir de vectores de características extraidos por cada parche; cada vector contiene la varianza, la curtosis y el sesgo calculados en la distribución de coeficientes obtenidos en método el realce descrito en la sección 2.3.1. Adicionalmente, en estos vectores se almacena el número de candidatos a microcalcificaciones segmentados en la sección 2.3.4 y sus respectivas etiquetas para las dos clases: tejido mamario sano y con microcalcificaciones mamarias agrupadas.
3. Resultados
Cada una de las imágenes mamográficas tiene la descripción del contorno del hallazgo marcado como anormal lo que permite validar el modelo. Además, la base de datos cuenta con información sobre el nivel de sutileza, en donde el primer nivel indica que las microcalcificaciones son sutilmente evidentes mientras que el quinto nivel son aquellas que se evidencian fácilmente y a simple vista.Para este trabajo, se extrajeron 86 parches: 43 con tejido mamario sano y 43 con microcalcificaciones agrupadas en distribuiciones segmentarias, lineales y ramificadas. Las imágenes mamográficas tienen un nivel de sutileza que va desde el segundo hasta el quinto nivel de sutileza.
La validación del modelo de clasificación se desarrolló bajo el esquema validación cruzada de 10 iteraciones (10-fold cross-validation). Las evaluación cuantitativa para la detección de regiones con microcalcificaciones mamarias se hizo empleando cuatro métricas: el área bajo la curva (Area Under the Curve, AUC, por sus siglas en inglés), precisión, sensibilidad y especificidad. En el cuadro I se muestra el desempeño de la clasificación binaria entre parches con tejido sano y con microcalcificaciones agrupadas. La mejor clasificación que se logró fue utilizando un SVM con kernel RBF (Radial Basis Function, RBF, por sus siglas en inglés).
Cuadro I: Resultados de la clasificación
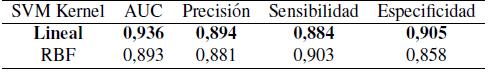
Cabe resaltar que el mejor desempeño en la clasificación se logró empleando el kernel lineal, logrando un área bajo la curva de 93,6% y una precisión de 89.4 %.
4. Discusión
Existen factores como: el realce de los ductos mamarios y regiones de tejido denso, la presencia de artefactos y errores en la captura de la imagen que aumentan el índice de falsos positivos ocasionando que se reduzca la especificidad del algoritmo. Por ejemplo, en el borde del tejido mamario la iluminación no es uniforme y presenta una alta variación en los niveles de gris que, con el método de realce produce la segmentación de algunos elementos que no son de interés, de modo que aumenta el número de falsos positivos. Esto se puede ver en la Figura 5.
Figura 5: (a) Imagen original del borde del tejido mamario. (b) Realce de la imagen original con el método propuesto. (c) Segmentación de la imagen. En la imagen c) se segmentan algunos elementos que no son microcalcificaciones aumentando el número de falsos positivos.
En algunas regiones del tejido mamario, la densidad es muy alta, presentando poca variación en los niveles de gris con respecto a las microcalcificaciones, causando que no haya realce de las microcalcificaciones, disminuyendo el acierto en las detecciones; lo anterior provoca una reducción en la sensibilidad de la prueba. Entre más bajo sea el nivel de sutileza menos evidentes son las microcalcificaciones, lo cual se puede ver en la Figura 6. Adicionalmente, con el método de realce propuesto y por causa de la eliminación subbanda, algunos posibles candidatos a microcalcificaciones desaparecen en la reconstrucción de la imagen, provocando que no sean segmentados, aumentando el número de falsos negativos.
Figura 6: (a) Imagen original que muestra el tejido denso con microcalcificaciones demarcadas en los círculos rojos. (b) Realce de la imagen original. (c) Algunas microcalcificaciones encerradas en círculos rojos en a) no son segmentadas
Los resultados son optimistas dado que se usa validación cruzada de 10 iteraciones (10-fold crossvalidation).Es preciso aclarar que para obtener resultados significativos, repetimos el experimento de clasificación veinte veces y obtuvimos un AUC con una media de 0;936 ? 0;084 para el kernel lineal y una media de 0; 8930 ? 0;0092 para el kernel RBF presentados en el cuadro I. Adicionalmente,la clasificación supera algunos de los trabajos mencionados en la literatura motivando la exploración de otras características. Finalmente, se recomienda para futuros trabajos explorar otras características morfológicas de las microcalcificaciones que no se tuvo en cuenta en este trabajo.
5. Conclusiones
En el enfoque presentado en este trabajo se tuvo en cuenta el tamaño y la distribución de microcalcificaciones,que en la evaluación clínica, son descriptores cuantitativos que comúnmente están asociados a la presencia de cáncer de mama en sus primeras etapas. Por otro lado, con el método de realce se logran desenmascarar algunas microcalcificaciones que en medio del tejido mamario denso no son evidentes a simple vista. En consecuencia, a través del enfoque propuesto se logra clasificar correctamente la mayoría de imagenes de tejido mamario con microcalcificaciones agrupadas ya sea en distribuciones segmentarias, lineales o ramificadas.
Acknowledgements
Agradecimientos
Los autores desean agradecer a la DDSM por facilitar la base de datos de las imágenes mamográficas. Igualmente al doctor Chris Rose por el software para la conversión de formato de las imágenes de la base de datos.
Referencias
License
From the edition of the V23N3 of year 2018 forward, the Creative Commons License "Attribution-Non-Commercial - No Derivative Works " is changed to the following:
Attribution - Non-Commercial - Share the same: this license allows others to distribute, remix, retouch, and create from your work in a non-commercial way, as long as they give you credit and license their new creations under the same conditions.








2.jpg)