DOI:
https://doi.org/10.14483/udistrital.jour.reving.2015.1.a04Published:
2015-04-07Issue:
Vol. 20 No. 1 (2015): January - JuneSection:
ArticleSegmentación y Conteo de las Líneas de la Nariz del Feto en Imágenes Ecográficas de las 11-13+6 Semanas de Gestación
Segmentation and nasal line counting in ultrasound fetal images at the 11-13+6 weeks of gestation
Keywords:
Homomorphic filtering, H-maxima transform, morphological reconstruction, geodesic dilation, eccentricity and clustering (en).Keywords:
Filtro homomórfico, transformada H-máxima, reconstrucción morfológica, dilatación geodésica, excentricidad, agrupamiento (es).Downloads
References
Kypros H. Nicolaides y Orlando Falcón, La ecografía de las 11- 13+6 semanas. Fetal Medicine Foundation, Londres, 2004.
K. T. a. Anjit and S. Rishidas, “Identification of nasal bone for the early detection of down syndrome using Back Propagation Neural Network,” 2011 International Conference on Communications and Signal Processing, 2011, pp. 136–140.
Sami Abdulla y Haidi Ibrahim, “Mathematical Equations for Homomorphic Filtering in Frequency Domain : A Literature Survey,” 2012 International Conference on Information and Knowledge Management (ICIKM), 2012, pp. 74–77.
Rafael. C. Gonzalez y Richard. E. Woods, Digital Image Processing, 3rd ed. Pearson International, 2009.
Nikhil J. Dhinagar y Mehmet Celenk, “Ultrasound Medical Image Enhancement and Segmentation Using Adaptive Homomorphic Filtering and Histogram Thresholding,” 2012 International Conference on Biomedical Engineering and Sciences, 2012, pp. 349–353.
Departamento de Electrónica y Automática - Facultad de Ingeniería - U.N.S.J Capítulo 3: Técnicas de realce de Imá-genesImágenes en Medicina (Bioingeniería), disponible en http://dea.unsj.edu.ar/imagenes/recursos/capitulo3.pdf
P. Soille, Morphological Images Analysis, Principles and Applications. 2nd ed, springer, Italia, 2003-2004.
Luc Vincent, “Morphological grayscale reconstruction in image analysis: applications and efficient algorithms”. IEEE Transactions on Image Processing, volumen 2, Número 2, 1993, pp.176-201.
Miguel Garre, Juan Cuadrado, Miguel A. Sicilia, Daniel Rodríguez y Ricardo Rejas, “Comparación de diferentes algoritmos de clustering en la estimación de coste en el desarrollo de software.” Revista Española de Innovación, Calidad e Ingeniería del Software, Volumen 3, Número 1, 2007, pp. 6–22.
Matteo Matteucci, A tutorial on Clustering algorithms: Fuzzy K-means, 2013, disponible en http://home.deib.polimi.it/matteucc/Clustering/tutorial_html/cmeans.html
Omer Demirkaya, Prasanna K. Sahoo, and Musa Hakan, Images Processing with MATLAB, Aplications in Medicine and Biology, CRC Press, 2009J. Jones, Networks, 10 de Mayo de 1991, disponible en http://www.atm.com.
Ranjan Maitra y Ivan P. Ramler, A k -mean-directions Algorithm for Fast Clustering of Data on the Sphere, disponible en http://www.public.iastate.edu/~maitra/papers/kmndirs.pdf
Bharati R.Jipkate y V.V.Gohokar, “A Comparative Analysis of Fuzzy C Means Clustering and K Means Clustering Algorithms”, International Journal Of Computational Engineering Research, Volumen 2, Número 3, May-June 2012, pp. 737–739.
Klaus D. Toennies, Guide to Medical Image Analysis. Springer, London, 2012.
Luigi Di Stefano y Andrea Bulgarelli, “A simple and efficient connected components labeling algorithm,” 10th International Conference on Image Analysis and Processing. IEEE Comput. Soc, 1999, pp. 322–327.
James D. Leeper, What is the difference between categorical, ordinal and interval variables?, 2014, disponible en http://www.ats.ucla.edu/stat/mult_pkg/whatstat/nominal_ordinal_interval.htm
Rajul Parikh, Annie Mathai, Shefali Parikh, G Chandra Sekhar, y Ravi Thomas, “Understanding and using sensitivity, specificity and predictive values”, Indian journal of ophthalmology, Volumen 56, Número 1, 2008, pp. 45–50.
How to Cite
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Download Citation
Segmentación y conteo de las líneas de la nariz del feto en imágenes ecográficas de las 11-13+6 semanas de gestación
Segmentation and nasal line counting in ultrasound fetal images at the 11-13+6 weeks of gestation
Angee Paola Ballesteros Maldonado, Bogotá – Colombia. apballesterosm@correo.udistrital.edu.co
Laura Catalina López Alza, Bogotá – Colombia. lclopeza@correo.udistrital.edu.co
Rodrido Javier Herrera García, Bogotá – Colombia. rherrera@udistrital.edu.co
Recibido: 19-06-2014. Modificado: 03-03-2015. Aceptado: 07-04-2015
Resumen
Este documento presenta un método para la segmentación de la nariz y el posterior conteo de las líneas correspondientes a la piel, el hueso nasal y la punta de la nariz en imágenes de ultrasonido fetales 2D del primer trimestre de gestación, de 11 a 13 semanas y 6 días (11-13+6 semanas). La presencia de las tres líneas del área nasal es una de las características que debe cumplir el feto en la ecografía para garantizar la confiabilidad de la medición del grosor translucencia nucal. Dicha medición se emplea para identificar la posible existencia de síndromes o anomalías cromosómicas. El método se aplica a la región correspondiente al corte sagital medio de la cabeza donde se han resaltado las regiones con mayor intensidad. La segmentación es jerárquica: primero, se identifica la región de la nariz a través de máximos regionales y alta excentricidad; posteriormente las líneas ya mencionadas son extraídas con agrupamiento basado en K- medias y morfología matemática.
Dada la incertidumbre entre las imágenes con respecto a posición fetal, resolución, nivel de ruido y rango dinámico, los resultados obtenidos van desde casos en los que no se encuentra la región de interés, hasta otros en los que son claramente identificables las líneas.
Palabras claves: filtro homomórfico, transformada H-máxima, reconstrucción morfológica, dilatación geodésica, excentricidad y agrupamiento.
Abstract
This document introduces a method for nasal segmentation and the subsequent counting of lines corresponding to the skin, nasal bone and the tip of the nose in 2D fetal ultrasound images of the first gestational trimester from 11 to 13 weeks and 6 days (11- 13+6 weeks). Presence of the three lines in the nasal region is one of the features that should have the fetus on ultrasound image to ensure the reliability of the measurement of fetal nuchal translucency thickness; this measurement is used to identify the possible existence of syndromes or chromosomal abnormalities. The method is applied to the mid-sagittal head plane where the hyperintense regions have been identified. The segmentation is hierarchical: first, the nose is identified by the combination of a regional maxima and high eccentricity detection algorithms; then the aforementioned lines are identified using K-means clustering and mathematical morphology.
Because of uncertainty between images with respect to fetal position, resolution, noise and dynamic range, the results ranging from cases in which the region of interest is not found, to those where the lines are clearly identifiable.
Key words: Homomorphic filtering, H-maxima transform, morphological reconstruction, geodesic dilation, eccentricity and clustering.
1. Introducción
En 1866, Langdon Down determinó como características comunes en las personas con trisomía 21 la hipotonía muscular, cara achatada y nariz pequeña [1]. Hacia los años noventa se determinó que la hipotonía muscular podría observarse en las ecografías del tercer mes de gestación como un incremento en el ancho de la translucencia nucal, que es una acumulación de líquido subcutáneo en la parte posterior del cuello del feto. Esta acumulación de líquido está relacionada además con otras anomalías cromosómicas, como la trisomía 13 y la 18, y se asocia también con anomalías mayores en el corazón y los grandes vasos. La valoración de imágenes ecográficas fetales es un procedimiento común en la práctica clínica, dado que el seguimiento del estado del feto en la gestación es fundamental para garantizar la salud de la madre y su hijo durante y después del embarazo.
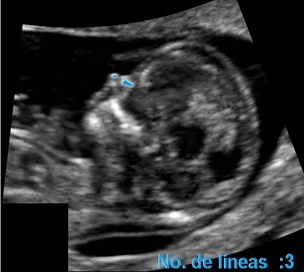
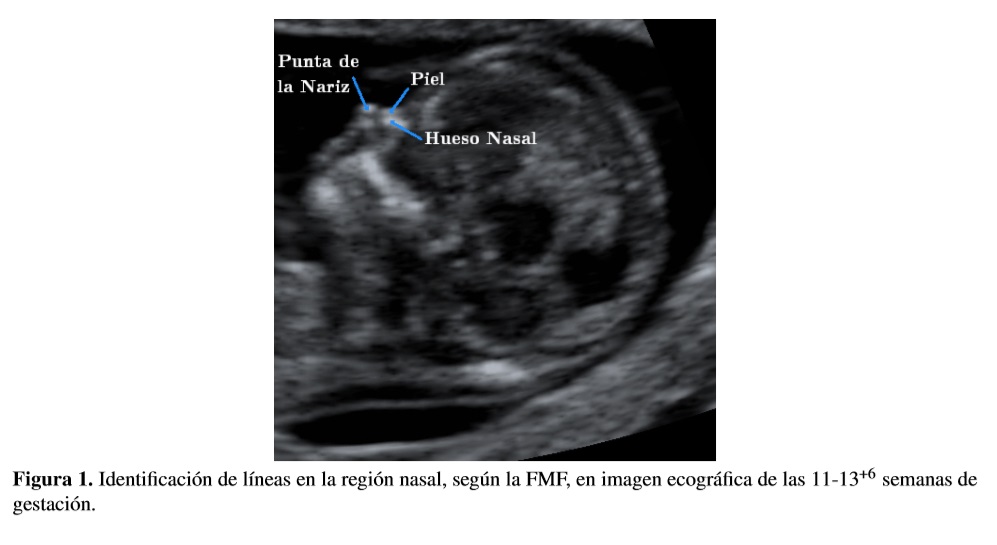
La confiabilidad de una correcta medición de la translucencia nucal en la ecografía depende de ciertas características que debe cumplir el feto en la imagen de ultrasonido. En consecuencia, se deben evaluar las imágenes tomadas por los especialistas. La presencia de tres líneas en la región nasal del feto es un parámetro utilizado en el proceso de auditoría de imágenes ecográficas de las 11-13+6 semanas de gestación correspondientes al primer trimestre, ya que determina un correcto plano sagital medio del perfil fetal. La línea superior representa la piel y la inferior, que es más gruesa y de mayor ecogenicidad, corresponde al hueso nasal. La tercera línea, casi en continuidad con la piel pero en un nivel más alto, es la punta de la nariz [1] (Figura 1).
En la parte anterior de la cabeza del feto, la nariz contiene píxeles de alta intensidad. Aun así, el ruido moteado de tipo multiplicativo (speckle) y manchas no comunes (como por ejemplo la presencia de la mano del feto, parte del útero de la madre o distorsiones de la imagen por movimiento del feto o densidad del tejido adiposo de la madre) consecuentes al proceso de adquisición de las imágenes son factores que generan errores en la identificación correcta del área nasal y en consecuencia en el conteo de las tres líneas mencionadas [2].
En el 2011, Anjit y Rishidas propusieron un método para la identificación del hueso nasal, con el que obtuvieron una tasa de detección del 88% y en el que usaron una definición preliminar de la región de interés, transformada watershed para la segmentación, transformada discreta de coseno para obtener los datos y redes neuronales [2]. Sin embargo, se requiere la selección de la región de interés por parte del experto, que aunque se respalda con su experiencia no deja de tener un elemento subjetivo. A diferencia del anterior, el método propuesto en este documento es completamente automático, lo que podría llegar a disminuir el tiempo de análisis y el componente de subjetividad que se presenta en la evaluación de las imágenes. Por consiguiente, es posible reducir las probabilidades de hacer mediciones incorrectas al tener certeza de que se cuenta con imágenes que cumplen con los criterios establecidos por la Fundación de Medicina Fetal (FMF) [1]. Además es aplicable a imágenes ecográficas con una alta incertidumbre entre ellas (posición fetal, resolución, nivel de ruido y rango dinámico). El método presentado no se plantea como una técnica de diagnóstico, sino como una herramienta que pueda emplear el especialista en la auditoría de las imágenes ecográficas.
La imagen de la cual parte el método propuesto corresponde a una segmentación previa de la región anterior de la cabeza del feto, y la extracción de la región de interés se basa en el empleo de máximos regionales y agrupamiento no supervisado.
El documento se encuentra organizado de la siguiente forma: La sección 2 describe el método utilizado. La sección 3 muestra resultados experimentales en 299 imágenes ecográficas. La sección 4 presenta la discusión de los resultados obtenidos en la sección 3. Finalmente las conclusiones son mostradas en la sección 5.
2. Metodología
Se propone un método para la detección de la región nasal y el conteo de las líneas que la componen. El método propuesto parte de una presegmentación de la cabeza del feto (Figura 4a). Puesto que el ruido y otras regiones que aparecen como resultado de la adquisición de la imagen son factores que introducen error en la identificación correcta del área nasal, se aplica un filtrado que atenúa el ruido y realza regiones hiperintensas (alta ecogenicidad) antes de la segmentación de esta región. El algoritmo de segmentación se basa en el empleo de máximos regionales y la evaluación de las características de dichos máximos (Figura 2).
2.1. Filtro Homomórfico
Debido a la adquisición de las imágenes, las regiones que deberían ser uniformes resultan ser más brillantes en algunas áreas y más oscuras en otras dependiendo de las condiciones de la paciente y el feto durante el examen con el ginecobstetra. Pueden encontrarse imágenes con estructuras de fácil diferenciación u otras con alto ruido como consecuencia de la cantidad de tejido adiposo de la madre o movimientos continuos del feto. Esta situación puede ser la causa de una clasificación errónea y en consecuencia de una inadecuada segmentación [3].
El filtro homomórfico puede ser usado para mejorar la apariencia de una imagen a través de la compresión simultánea del rango de intensidades y el aumento de contraste [4]. Para un filtro homomórfico, una imagen f se define teóricamente como:
Donde i(x,y) es el componente de iluminación, principal responsable del rango dinámico, y r(x,y) es la reflectancia, principal responsable del contraste local [5].
Se calcula el logaritmo de la función:
Posteriormente se halla la transformada de Fourier para operar en el dominio de la frecuencia:
La función de filtrado homomórfico puede ser construida usando un filtro de alto impulso (pasa alto) Butterworth de primer orden:
Donde D0, γL, γH son constantes positivas y Du,v es la distancia entre un punto (u,v) en el dominio de la frecuencia y el centro del rectángulo de frecuencias:
Entonces es posible filtrar Z(u,v) con Hu,v
Que en el dominio del espacio estaría dado por:
Dado que aplicamos logaritmo natural en un principio, debemos al resultado final hallar su exponencial:
Estos son los componentes que contienen la información del nivel de gris promedio y las variaciones abruptas presentes en bordes y fronteras entre objetos, respectivamente [6]. Las bajas frecuencias de la transformada de Fourier del logaritmo de una imagen se relacionan con el rango dinámico, y las altas frecuencias se relacionan con el contraste local.
Las zonas homogéneas en la imagen dan lugar a que la energía del espectro esté concentrada mayoritariamente en las bajas frecuencias [4]. En las ecografías, las regiones de interés son homogéneas por tal motivo si γH < 1 y γL > 1, el filtro tiende a resaltarlas.
Para la segmentación y partiendo del hecho de que se han resaltado las regiones de interés, se emplea la transformada máxima extendida, que permite identificar grupos de píxeles que tienen un valor de intensidad significativamente más alto que el de sus vecinos.
2.2. Transformada máxima extendida
La región nasal es un objeto que se distingue fácilmente del resto de la imagen puesto que es hiperintensa, por tanto, la imagen máxima es una importante característica morfológica porque permite usar esta singularidad para seleccionar objetos relevantes en las imágenes.
Latransformadamáximaextendidaesunatransformacióndelgrupodetransformadasgeodésicas, compuesta a su vez por la transformada H-máxima, que emplea el concepto de reconstrucción morfológica a partir de la transformada básica de dilatación geodésica.
2.2.1. Dilatación geodésica
En la dilatación geodésica se trabaja con 2 imágenes: una imagen marcador f y una imagen máscara g. Ambas imágenes tienen el mismo dominio (Df = Dg) y la imagen máscara debe ser mayor o igual que la imagen marcador desde el punto de vista de sus niveles de gris (f ≤ g). La imagen marcador es dilatada por un elemento estructurante isotrópico, y posteriormente es forzada a permanecer por debajo de la imagen máscara. La imagen máscara actúa como un límite a la dilatación de la imagen marcador.
En otras palabras, la dilatación geodésica de tamaño 1 está definida como el punto mínimo entre la imagen máscara y la dilatación elemental δg(1) de la imagen marcador [7]:
La dilatación geodésica de tamaño n puede ser obtenida por n iteraciones de dilataciones geodésicas elementales [8]:
2.2.2. Reconstrucción morfológica
La reconstrucción morfológica de una imagen máscara g a partir de una imagen marcador f se basa en la convergencia de las transformaciones geodésicas de imágenes delimitadas después de un número finito de iteraciones, y está definida como la dilatación geodésica de f con respecto a g iterativa hasta la estabilidad. Se denota por Rgδ(f):
Donde i es tal que δg(i)(f) = δg(i+1)(f).Es decir, i es tal que las transformaciones geodésicas convergen.
2.2.3. Transformada H-Máxima
La transformada H-máxima suprime todos los máximos cuya altura es menor o igual al del valor de umbral h dado. Esto se consigue realizando la reconstrucción por dilatación de f desde f −h:
Donde Rf (f −h) es la reconstrucción morfológica por dilatación de la imagen desde ( f −h) [7].
2.2.4. Transformada máxima extendida
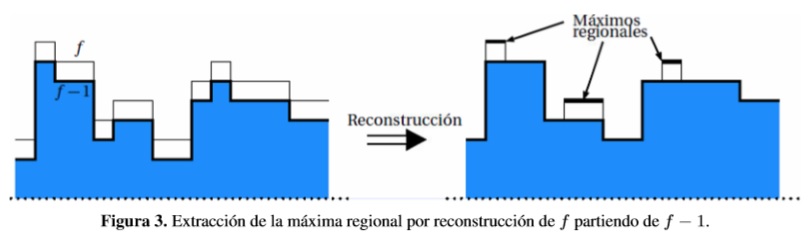
Un máximo regional M de una imagen f es un componente de píxeles conectados con una intensidad t donde el borde externo tiene un valor estrictamente menor que t [7]. Todos los píxeles pertenecientes a un máximo regional son un máximo local, pero no de forma opuesta [8].
La transformada H-máxima (HMAX) filtra los máximos regionales empleando un criterio de contraste.La transformada máxima es extendida si un máximo original, es decir un máximo regional de la imagen no reconstruida, todavía pertenece a un máximo regional de la imagen reconstruida.
La transformada máxima extendida (EMAX) está definida como la máxima regional de la correspondiente transformación (HMAX):
La máxima regional para una señal de una dimensión y h = 1 se observa en la Figura 3 [8].
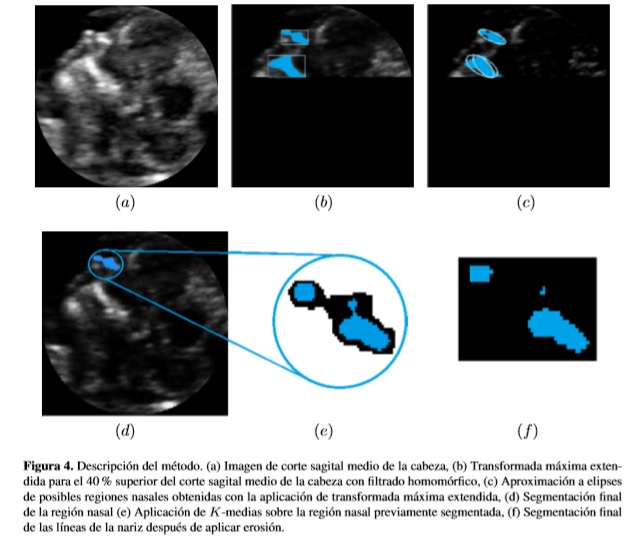
En la Figura 4b se presenta el resultado al aplicar transformada máxima extendida en el 40% de las imágenes ecográficas.
Dada la incertidumbre entre las imágenes y el ruido speckle presente en ellas,la transformada máxima extendida selecciona, en algunos casos, regiones con áreas significativas, debido a la dificultad para diferenciar los máximos regionales. Por tanto, se realizó un estimativo del área de la región de la nariz respecto a la cabeza del feto, resultando en la mayoría de casos inferior a 1,5%. Cuando la región tiene un valor superior, se aplica a ella la transformada máxima extendida con el fin de dividirla en secciones más pequeñas que tienen mayor probabilidad de corresponder a la región de la nariz. En los demás casos, aquellos en los que se selecciona regiones con áreas no significativas (inferiores a 1,5%), se toman estas como probables regiones de la nariz.
Las aproximaciones de las regiones a elipses permiten la obtención de medidas de las mismas, que pueden emplearse como patrones de selección. La excentricidad, por ejemplo, permitediferenciarelementosalargadosdecirculares.Aplicadoaimágenesecográficas,laregión de la nariz puede entonces aproximarse a una elipse con alta excentricidad, como se observa en la Figura 4c. Sin embargo, deben excluirse los casos en los que el eje principal de la elipse es superior al 25% de la media de las dimensiones de la imagen, dado que pueden confundirse con otras zonas de mayor tamaño y con formas similares, tal como el maxilar superior, como se observa en la Figura 4d.
2.3. Agrupamiento por K-medias
El agrupamiento K-medias es la representación de grupos a través de la media (o media ponderada) de sus puntos asignando a cada dato un centroide o pertenencia binaria [9], [10]. El uso de este método permite la segmentación de las líneas de la nariz representadas como pequeños grupos de píxeles en la región nasal.
El algoritmo requiere la especificación del número de grupos que se desean, encontrando una división óptima tal que la suma de las distancias euclidianas cuadradas entre un punto y su centroide se reduce al mínimo a nivel local; en ese contexto, la segmentación de imágenes puede modelarse como la identificación de grupos de píxeles [11], [12].
El algoritmo minimiza la suma de las varianzas de cada grupo, definida como la suma de los cuadrados de las diferencias entre los puntos y sus respectivos centroides, como se presenta en la Ecuación 14.
Donde vij es la i-ésima muestra de la j-ésima clase Kj y µj el centro del j-ésimo grupo definido como la media de vi ∈ Kj y ||vij − µj|| es la norma de vij − µj [11].
El resultado del agrupamiento K-medias depende de la inicialización de los centroides, así que se recomienda seleccionarlos aleatoriamente y lo más lejos posible el uno del otro con el fin de que el algoritmo obtenga el mínimo local óptimo de la Ecuación 14 [13], [14].
Para la segmentación de las líneas de la nariz se definen dos grupos los cuales corresponden a tejido óseo (mayor ecogenicidad) y a tejidos blandos de la región. La identificación del tejido óseo corresponde a la región a evaluar posteriormente. El resultado de aplicar K-medias a la región nasal se observa en la Figura 4e. Finalmente se emplea erosión para realizar su diferenciación (para detalles de la erosión ver [4]). El resultado final se observa en la Figura 4f.
2.4. Etiquetado por componentes conexos
Para el análisis de los objetos en las imágenes binarias, estos generalmente son extraídos empleando la operación de etiquetado por componentes conectados, que consiste en la asignación de una etiqueta única a cada componente [15]. Generalmente dichas etiquetas son enteros positivos.
Dos píxeles P y Q están conectados si existe un camino de píxeles (p0,p1,...,pn) tal que p0 = P, pn = Q y∀1 ≤ i ≤ n, pi−1 y pi son vecinos adyacentes. Por lo tanto, la definición de componentes conectados se basa en el vecindario del píxel. Si tiene 4-vecinos, C es un componente con conectividad 4, por otro lado si tiene 8 vecinos, C tiene conectividad 8.
Por medio de este etiquetado y usando conectividad 8 se realiza entonces el conteo de las líneas nasales previamente segmentadas.
2.5. Validación
En la evaluación de la segmentación de la región nasal se mide la tasa de detección, la cual se define como la relación entre casos detectados y el total de casos analizados. Por otro lado, puesto que el número de líneas de la nariz es una variable categórica, es decir que indica categorías o es etiquetada numéricamente (1,2 o 3 líneas), y no existe un ordenamiento intrínseco de sus niveles [16], no es adecuado hallar medidas como el promedio; son más convenientes otras como el valor predictivo positivo.
El valor predictivo positivo (V PP) es una medida porcentual que indica cuántos resultados positivos son realmente positivos, y que se respalda con la región identificada por el experto, la cual asumimos como el “gold standard” o patrón de referencia. Si el valor es alto (cercano a 100%), advierte que el algoritmo propuesto entrega resultados acertados [17].
En términos de la detección correcta de las líneas de la nariz, se calcula como la relación de la cantidad de líneas encontradas y que corresponden al patrón de referencia (verdaderos positivos), y el total de las líneas halladas (verdaderos positivos (V P) más falsos positivos (FP) (Ecuación 15):
Adicionalmente se mide el porcentaje de error del número de líneas total por categoría, como se muestra en la Ecuación 16.
3. Resultados
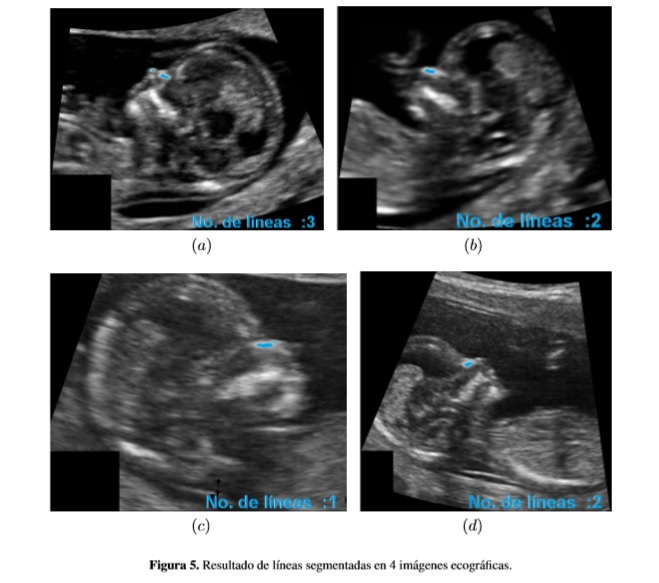
La base de datos usada en la validación de este método está compuesta por un conjunto de 299 imágenes ecográficas de diferentes pacientes. La adquisición de las imágenes fue realizada a través del ecógrafo Voluson E6 con una resolución de 800×600 píxeles en espacio RGB. Las Figuras 5a y 6b muestran el ejemplo de una correcta segmentación; en la Figura 5c se aprecia el ejemplo de la confusión de líneas debido a la cercanía de la piel y la punta de la nariz. Finalmente, la Figura 5d muestra un ejemplo de cuando alguna línea no es hallada.
Con respecto a la región de la nariz para las 299 imágenes se identificó correctamente la región nasal en 187, es decir en 62,54% de las imágenes.
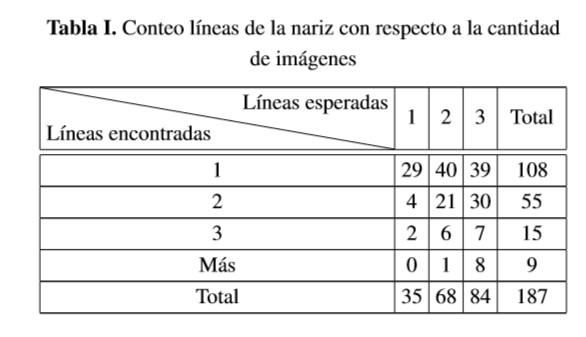
La Tabla I indica los resultados de la cantidad de imágenes relacionadas con el número de líneas identificadas, y la Tabla II relaciona la cantidad de VP y FP del total de líneas, para las 187 imágenes en las que la región nasal fue correctamente detectada.
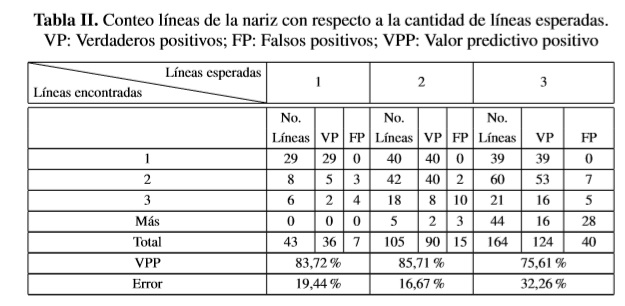
La tasa de detección de la región de la nariz fue de 62,54% para imágenes en las cuales se encontraba el área nasal. En cuanto a las líneas de la nariz, el mejor valor predictivo positivo obtenido fue de 85,71% con error de 16,67% para imágenes con 2 líneas; y el peor, de 75,61% con error de 32,26% para imágenes con 3 líneas.
4. Discusión
El análisis realizado sobre la totalidad de las imágenes permitió establecer características comunes como el estimativo del área de la región nasal en 1,5% respecto a la cabeza del feto y la similitud de esta a una elipse, que facilitan la adaptación del modelo a cada ecografía fetal y en consecuencia contrarresta en gran parte la incertidumbre entre imágenes. De cualquier manera,esta última tuvo implicaciones directas en los resultados obtenidos, ya que apesar de que el método planteado es automáticamente ajustable a cada imagen, factores como el ruido, debido a elementos como la cantidad de tejido adiposo materno o movimientos continuos del feto, dificultan la segmentación.
Los resultados obtenidos en tasa de detección de la región de la nariz crean la necesidad de buscar nuevas alternativas para aumentar el porcentaje de detección. En cuanto a los resultados obtenidos en el número de líneas de la nariz se debe considerar la existencia de trabajos con resultados de mayor valor porcentual, como por ejemplo el trabajo de Anjit y Rishidas que presenta una tasa de detección del 88%, pero que fueron precedidos por una preselección de las imágenes. En este trabajo, a diferencia, no se realizó ninguna preselección puesto que se buscó desarrollar un método como punto de partida para la extracción de características de cualquier imagen ecográfica de las 11-13+6 semanas de gestación. Es de anotar que una preselección realizada por un experto en imágenes ecográficas fetales descarta en promedio un 90-95% de las imágenes tomadas por otros especialistas.
Por otra parte, la cercanía que presenta la piel y la punta de la nariz hace que se confundan tales líneas y aumente el error en su conteo. Esto se refleja en los resultados que muestran la menor tasa de detección en las imágenes con tres líneas y la mayor tasa de detección en las que contienen dos líneas. Adicionalmente, el ruido (spekle) afecta la detección, en tanto que al ser ruido moteado se confunde con las líneas y da lugar a los falsos positivos. Asímismo,la atenuación en las variaciones de contraste añadidas por el ruido genera la confusión de unas líneas con otras de forma que el método llega a idenficar dos líneas diferentes como una sola.
Por otro lado, el proceso jerárquico desarrollado implica subordinación de los resultados de una etapa a la anterior. Por lo tanto, los resultados finales correspondientes al número de líneas dependen directamente de la selección adecuada de la región nasal. Esto debido a que si la región no se identificó en su totalidad, se disminuye el número de líneas que se segmentan. Las variaciones de contraste son determinantes en la segmentación adecuada de la región nasal y las líneas de la nariz.
5. Conclusiones
El método propuesto establece un punto de partida para la investigación dedicada a la extracción automática de características, útiles en la validación de imágenes ecográficas de las 11-13+6 semanas de gestación. Debido a que este trabajo es una incursión inicial en la investigación de la selección de parámetros de este tipo de imágenes, no es un resultado definitivo. Sin embargo,a futuro puede llegar a ser una herramienta de ayuda para la auditoría de imágenes tomadas por los especialistas, lo que permitiría disminuir la subjetividad en la evaluación de las imágenes. En el proceso de auditoría de las imágenes son eliminadas un promedio de 90 a 95% por no ser idóneas para el análisis por parte del especialista. Como trabajo futuro se sugiere el uso de otros métodos de procesamiento que permitan balancear la variación de intensidades de manera homogénea, con el fin de mejorar la selección de máximos regionales, lo que aumentaría la tasa de detección del área nasal. Por otro lado, modelos de aprendizaje no supervisado, tales como la máquina de soporte de vectores o support vector machine (SVM), podrían realizar una clasificación más detallada de la cantidad de líneas de la nariz; adicionalmente incluirían el parámetro de 0 líneas que no se evalúa en este trabajo como consecuencia de la naturaleza de los métodos utilizados. Así mismo y dado que el especialista al entregar el “gold standard” o criterio de comparación solo entrega el número total de líneas, en un trabajo futuro se podría evaluar que la l´Inea encontrada corresponda claramente a la punta de la nariz, el hueso nasal o piel, sin embargo no hay características de forma que las diferencien. De igual manera, dedicar un trabajo al modelamiento del ruido speckle sería un punto de partida para la mejora notable de los resultados.
Agradecimientos: Los autores agradecen a la Clínica de Obstetricia y Ginecología Ltda y al Dr. Jaime Arenas Gamboa por sus invaluables sugerencias y por el tiempo dedicado en la recolección de la base de datos necesaria para este estudio.
Referencias
-
Kypros H. Nicolaides y Orlando Falcón, La ecografía de las 11- 13+6 semanas. Fetal Medicine Foundation, Londres, 2004.
-
K. T. A. Anjit y S. Rishidas, “Identification of nasal bone for the early detection of down syndrome using Back Propagation Neural Network, ” 2011 International Conference on Communications and Signal Processing, 2011, pp. 136–140.
-
Sami Abdulla y Haidi Ibrahim, “Mathematical Equations for Homomorphic Filtering in Frequency Domain : A Literature Survey, ” 2012 International Conference on Information and Knowledge Management (ICIKM), 2012, pp. 74–77.
-
Rafael. C. Gonzalez y Richard. E. Woods, Digital Image Processing, 3rd ed. Pearson International, 2009.
-
Nikhil J. Dhinagar y Mehmet Celenk, “ Ultrasound Medical Image Enhancementand Segmentation Using Adaptive Homomorphic Filteringand Histogram Thresholding, ” 2012 International Conference on Biomedical Engineering and Sciences, 2012, pp. 349–353.
-
Departamento de Electrónica y Automática - Facultad de Ingeniería - U.N.S.J Capítulo 3: Técnicas de realce de Imágenes en Medicina (Bioingeniería), disponible en http://dea.unsj.edu.ar/imagenes/recursos/capitulo3.pdf
-
P. Soille, Morphological Images Analysis, Principles and Applications. 2nd ed, Springer, Italia, 2003-2004.
-
Luc Vincent, “Morphological grayscale reconstruction in imagean alysis : applications and efficient algorithms”. IEEE Transactions on Image Processing, volumen 2, Número 2, 1993, pp.176-201.
-
Miguel Garre, Juan Cuadrado, Miguel A. Sicilia, Daniel Rodríguez y Ricardo Rejas,“ Comparación de diferentes algoritmos de clustering en la estimación de coste en el desarrollo de software.” Revista Española de Innovación, Calidad e Ingeniería del Software, Volumen 3, Número 1, 2007, pp. 6–22.
-
Matteo Matteucci, A tutorial on Clustering algorithms: Fuzzy K-means, 2013, disponible en http://home.deib.polimi.it/matteucc/Clustering/tutorial html/cmeans.html
-
Omer Demirkaya, Prasanna K. Sahoo, and Musa Hakan, Images Processing with MATLAB, Aplications in Medicine and Biology, CRC Press, 2009J. Jones, Networks, 10 de mayo de 1991, disponible en http://www.atm.com.
-
Ranjan Maitra y Ivan P. Ramler, A k-mean-directions Algorithm for Fast Clustering of Data on the Sphere, disponible en http://www.public.iastate.edu/ maitra/papers/kmndirs.pdf
-
Bharati R. Jipkate y V.V.Gohokar, “A Comparative Analysis of Fuzzy C Means Clustering and K Means Clustering Algorithms”, International Journal Of Computational Engineering Research, Volumen 2, Número 3, May-June 2012, pp. 737–739.
-
Klaus D. Toennies, Guide to Medical Image Analysis. Springer, London, 2012
-
Luigi Di Stefano y Andrea Bulgarelli, “A simple and efficient connected components labeling algorithm,” 10th International Conference on Image Analysis and Processing. IEEE Comput. Soc, 1999, pp. 322–327.
-
James D. Leeper, What is the difference between categorical, ordinal and interval variables?, 2014, disponible en http://www.ats.ucla.edu/stat/mult pkg/whatstat/nominal ordinal interval.\htm
-
Rajul Parikh, Annie Mathai, Shefali Parikh, G Chandra Sekhar, y Ravi Thomas, “Understanding and using sensitivity, specificity and predictive values”, Indian Journal of Ophthalmology, Volumen 56, Número 1, 2008, pp. 45–50.
Angee Paola Ballesteros Maldonado
Nació en Bogotá, Colombia. Es ingeniera electrónica de la Universidad Distrital Francisco José de Caldas, de Bogotá, Colombia. Sus áreas de interés son el procesamiento digital de imágenes, la inteligencia computacional y el control.
e-mail: apballesterosm@correo.udistrital.edu.co
Laura Catalina López Alza
Nació en Bogotá, Colombia. Es ingeniera electrónica de la Universidad Distrital Francisco José de Caldas, de Bogotá, Colombia. Sus áreas de interés son el procesamiento digital de imágenes, la inteligencia computacional y el control.
e-mail: lclopeza@correo.udistrital.edu.co
Rodrigo Javier Herrera García
Nació en Bogotá Colombia. Es ingeniero electrónico de la Universidad Distrital, de Bogotá, Colombia. Obtuvo su título de Maestría en Ciencias de la Información y las Comunicaciones en la Universidad Distrital Francisco José de Caldas de Bogotá, Colombia. Se desempeñó como docente universitario desde 1988 en las Universidades Santo Tomas de Aquino y Distrital de Bogotá en las áreas de sistemas digitales y electrónica analógica. A partir de 1997 se vinculó como profesor de tiempo completo en la Facultad de Ingeniería de la Universidad Distrital Francisco José de Caldas.
Actualmente se desempeña como profesor en el área de procesamiento de señales en la Universidad Distrital Francisco José de Caldas de Bogotá, Colombia, y es director del grupo de investigación, desarrollo y aplicaciones en señales IDEAS-UD, donde realiza estudios sobre procesamiento digital de imágenes.
e-mail: rherrera@udistrital.edu.co

Este trabajo está autorizado por una Licencia Attribution-NonCommercial-NoDerivs CC BY-NC-ND.
License
From the edition of the V23N3 of year 2018 forward, the Creative Commons License "Attribution-Non-Commercial - No Derivative Works " is changed to the following:
Attribution - Non-Commercial - Share the same: this license allows others to distribute, remix, retouch, and create from your work in a non-commercial way, as long as they give you credit and license their new creations under the same conditions.




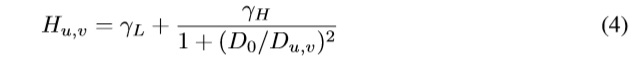
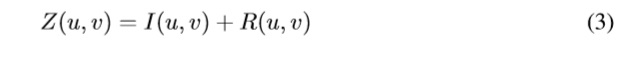
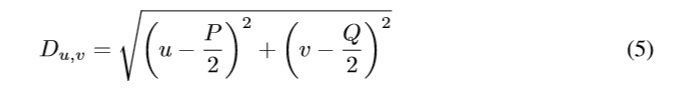
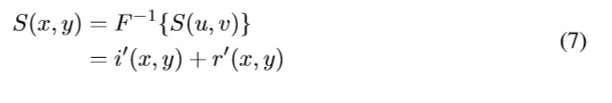
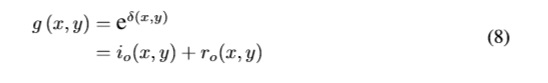

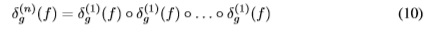


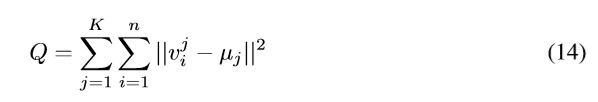





2.jpg)